Tema 8 Regresión Lineal
8.1 Regresión lineal simple
El problema de regresión consiste en hallar la mejor relación funcional entre dos variables \(X\) e \(Y\).
Más concretamente, dada una muestra de las dos variables \(X,Y\), \((x_i,y_i)_{i=1,2,\ldots,n}\), queremos estudiar cómo depende el valor de \(Y\) en función del valor de \(X\).
La variable aleatoria \(Y\) es la variable dependiente o de respuesta.
La variable (no necesariamente aleatoria) \(X\) es la variable de control, independiente o de regresión. Pensemos por ejemplo, en un experimento donde la variable \(X\) es la que controla el experimentador y la variable \(Y\) es el valor que se obtiene del experimento.
El problema de regresión es encontrar la mejor relación funcional que explique la variable \(Y\) conocidas las observaciones de la variable \(X\).
Si dicha relación funcional es una recta, \(Y=\beta_0 +\beta_1 x\), la regresión se denomina regresión lineal.
En la regresión lineal, se hace la suposición siguiente: \[ \mu_{Y|x}=\beta_0+\beta_1 x, \] dónde \(\mu_{Y|x}\) es el valor esperado de la variable aleatoria \(Y\) cuando la variable \(X\) vale \(x\). Dicho valor esperado es una función lineal de \(X\) con término independiente \(\beta_0\) y pendiente \(\beta_1\). Dichos valores son dos parámetros que tendremos que estimar.
Las estimaciones de \(\beta_0\) y \(\beta_1\) se llaman \(b_0\) y \(b_1\), respectivamente y se tienen que realizar a partir de la muestra \((x_i,y_i)_{i=1,2,\ldots,n}\).
Una vez halladas las estimaciones \(b_0\) y \(b_1\), obtendremos la recta de regresión para nuestra muestra: \[ \widehat{y}=b_0+b_1 x, \] que dado un valor \(x_0\) de \(X\), estimará el valor \(\widehat{y}_0=b_0+b_1 x_0\) de la variable \(Y\).
8.1.1 Mínimos cuadrados
Vamos a explicar el método para hallar las estimaciones \(b_0\) y \(b_1\).
Dicho método se denomina método de los mínimos cuadrados.
Dada una observación cualquiera de la muestra, \((x_i,y_i)\), podremos separar la componente \(y_i\) como la suma de su valor predicho por el modelo y el error cometido: \[ y_i=\beta_0+\beta_1 x_i+ \varepsilon_i\Rightarrow \varepsilon_i=y_i-(\beta_0+\beta_1 x_i). \] Llamamos error cuadrático teórico de este modelo a la suma al cuadrado de todos los errores cometidos por los valores de la muestra: \[ SS_\varepsilon=\sum_{i=1}^n \varepsilon_i^2=\sum_{i=1}^n (y_i-\beta_0-\beta_1 x_i)^2. \]
La regresión lineal por mínimos cuadrados consiste en hallar los estimadores \(b_0\) y \(b_1\) de \(\beta_0\) y \(\beta_1\) que minimicen dicho error cuadrático teórico \(SS_\varepsilon\).
Para hallar el mínimo del error cuadrático teórico, \((b_0,b_1)\), hay que derivar respecto las variables \(\beta_0\) y \(\beta_1\) e igualar a \(0\) dichas derivadas: \[ \begin{array}{ll} \dfrac{\partial SS_\varepsilon}{\partial \beta_0}_{|\beta_0=b_0,\beta_1=b_1}=&-2\sum\limits_{i=1}^n (y_i -b_0-b_1 x_i)=0,\\[1ex] \dfrac{\partial SS_\varepsilon}{\partial \beta_1}_{|\beta_0=b_0,\beta_1=b_1}=&-2\sum\limits_{i=1}^n (y_i -b_0-b_1 x_i) x_i =0. \end{array} \]
La solución del sistema anterior es: \[ \begin{array}{rl} b_1& \displaystyle=\frac{n \sum\limits_{i=1}^n x_i y_i-\sum\limits_{i=1}^n x_i\sum\limits_{i=1}^n y_i} {n\sum\limits_{i=1}^n x_i^2-(\sum\limits_{i=1}^n x_i)^2},\\ b_0& \displaystyle=\frac{\sum\limits_{i=1}^n y_i -b_1 \sum\limits_{i=1}^n x_i}{n}. \end{array} \]
Vamos a escribir las estimaciones \(b_0\) y \(b_1\) obtenidas anteriormente en función de las medias, varianzas y covarianza de la muestra de valores \((x_i,y_i)\).
Sean entonces \[ \overline{x}=\frac1{n}\sum\limits_{i=1}^n x_i, \quad \overline{y}=\frac1{n} \sum\limits_{i=1}^n y_i, \] las medias de las componentes \(x\) e \(y\) de los valores de la muestra y sean
\[ \begin{array}{rl} \tilde{s}_x^2 &\displaystyle =\frac1{n-1}\sum_{i=1}^n (x_i-\overline{x})^2 =\frac{n}{n-1}\left(\frac1{n}\Big(\sum_{i=1}^n x_i^2\Big) -\overline{x}^2\right),\\ \tilde{s}_y^2 &\displaystyle =\frac1{n-1}\sum_{i=1}^n (y_i-\overline{y})^2 =\frac{n}{n-1}\left(\frac1{n}\Big(\sum_{i=1}^n y_i^2\Big) -\overline{y}^2\right),\\ \tilde{s}_{xy} &\displaystyle =\frac1{n-1}\sum_{i=1}^n (x_i-\overline{x}) (y_i-\overline{y}) =\frac{n}{n-1}\left(\frac1{n}\Big(\sum_{i=1}^n x_i y_i\Big)-\overline{x}\cdot\overline{y}\right), \end{array} \] las varianzas y la covarianza de la muestra \((x_i,y_i)\), \(i=1,\ldots,n\).
Las estimaciones \(b_0\) y \(b_1\) se pueden reescribir de la forma siguiente:
Ejercicio
Se deja como ejercicio la demostración del teorema anterior a partir de la expresión hallada anteriormente.
Dado un valor \(x\) de la variable \(X\), llamaremos \(\widehat{y}\) a la expresión \(\widehat{y}=b_0+b_1x\) al valor estimado de \(Y\) cuando \(X=x\).
Dada una observación \((x_i,y_i)\), llamaremos error de la observación \(e_i\) a la expresión \(e_i=y_i-\widehat{y}_i={y}_i-b_0-b_1x_i\).
Ejemplo
En un experimento donde se quería estudiar la asociación entre consumo de sal y presión arterial, se asignó aleatoriamente a algunos individuos una cantidad diaria constante de sal en su dieta, y al cabo de un mes se los midió la tensión arterial media. Algunos resultados fueron los siguientes:
| \(X\) (sal, en g) | \(Y\) (Presión, en mm de Hg) |
|---|---|
| 1.8 | 100 |
| 2.2 | 98 |
| 3.5 | 110 |
| 4.0 | 110 |
| 4.3 | 112 |
| 5.0 | 120 |
Vamos a hallar la recta de regresión lineal por mínimos cuadrados de \(Y\) en función de \(X\).
En primer lugar calculamos las medias, varianzas y covarianza de la muestra de datos:
## [1] 3.467## [1] 108.3## [1] 1.543## [1] 66.27## [1] 9.773Los estimadores \(b_0\) y \(b_1\) serán:
## [1] 6.335## [1] 86.37La recta de regresión será, en este caso: \(\widehat{y} = 86.3708+6.3354\cdot x\).
8.1.2 Recta de regresión en R
Para hallar la recta de regresión en R hay que usar la función lm:
##
## Call:
## lm(formula = tensión ~ sal)
##
## Coefficients:
## (Intercept) sal
## 86.37 6.34La recta de regresión hallada por el método de los mínimos cuadrados verifica las propiedades siguientes:
La recta de regresión pasa por el vector medio \((\overline{x},\overline{y})\) de nuestra muestra de datos \((x_i,y_i)\), \(i=1,\ldots,n\): \[ \overline{y}=b_0+b_1 \overline{x}. \]
La media de los valores estimados a partir de la recta de regresión es igual a la media de los observados \(y_i\), \(\overline{y}\). Es decir: \[ \overline{\widehat{y}}=\frac1{n}\sum_{i=1}^n\widehat{y}_i =\frac1{n}\sum_{i=1}^n(b_0+b_1x_i)= b_0+b_1 \overline{x}=\overline{y}. \]
Los errores \((e_i)_{i=1,\ldots,n}\) tienen media 0: \[ \overline{e} =\frac1{n}\sum_{i=1}^n e_i =\frac1{n}\sum_{i=1}^n (y_i-b_0-b_1x) =\frac1{n}\sum_{i=1}^n (y_i-\widehat{y}_i) =0. \]
Llamaremos suma de cuadrados de los errores a la cantidad siguiente: \(\displaystyle SS_E=\sum_{i=1}^{n} e^2_i.\)
Usando que los errores \((e_i)_{i=1,\ldots,n}\) tienen media \(0\), su varianza será: \[ s_e^2=\frac1{n}\Big(\sum_{i=1}^{n} e^2_i\Big)-\overline{e}^2=\frac{SS_E}{n}-0=\frac{SS_E}{n}. \]
Definimos las variables aleatorias \(E_{x_i}\) como \(E_{x_i}=y_i-b_0-b_1\cdot x_i\) donde \((x_i,y_i)\) es un valor de la muestra y \(b_0\) y \(b_1\) son los estimadores obtenidos por el método de los mínimos cuadrados. Entonces,
- \(b_0\) y son \(b_1\) los estimadores lineales no sesgados óptimos (más eficientes) de \(\beta_0\) y \(\beta_1\), respectivamente.
y un estimador no sesgado de \(\sigma_E^2\) es el siguiente: \(S^2=\frac{SS_E}{n-2}\).
Si, además, las variables aleatorias error \(E_{x_i}\) son normales, entonces \(b_0\) y son \(b_1\) los estimadores máximo verosímiles de \(\beta_0\) y \(\beta_1\), respectivamente.
Ejemplo
Comprobemos las propiedades para los datos del ejemplo anterior:
- La recta de regresión pasa por el vector medio \((\overline{x},\overline{y})\):
## [1] 0- La media de los valores estimados a partir de la recta de regresión es igual a la media de los observados \(y_i\), \(\overline{y}\).
## [1] 0La estimación de la varianza para los datos del ejemplo anterior es la siguiente:
errores=tensión.estimada-tensión
SSE = sum(errores^2)
n=length(sal)
(estimación.varianza = SSE/(n-2))## [1] 5.436Entonces tenemos que el valor aproximado o estimado de \(\sigma_E^2\) es 5.4365.
8.1.3 Coeficiente de determinación
Llegados a este punto, nos preguntamos lo efectiva que es la recta de regresión.
Es decir, cómo medir si la aproximación hallada \(\widehat{y}=b_0+b_1 x\) a la nube de puntos \((x_i,y_i),\ i=1,\ldots,n\) ha sido suficientemente buena.
Una forma de realizar dicha medición es a través del coeficiente de determinación \(R^2\) que estima cuánta variabilidad de los valores \(y_i\) heredan los valores estimados \(\widehat{y}_i\).
Para ver su definición, necesitamos introducir las variabilidades siguientes:
Variabilidad total o suma total de cuadrados: \(SS_T =\sum\limits_{i=1}^n(y_i-\overline{y})^2 = (n-1)\cdot \tilde{s}_y^2.\)
Variabilidad de la regresión o suma de cuadrados de la regresión: \(SS_R=\sum\limits_{i=1}^n(\widehat{y}_i-\overline{y})^2=(n-1)\cdot \tilde{s}_{\widehat{y}}^2.\)
Variabilidad del error o suma de cuadrados del error: \(SS_E=\sum\limits_{i=1}^n(y_i-\widehat{y}_i)^2=(n-1)\cdot \tilde{s}_e^2.\)
El teorema siguiente nos relaciona las variabilidades anteriores:
Entonces, cuántas más “próximas” estén las variabilidades \(SS_T\) y \(SS_R\), o, si se quiere, \(\tilde{s}^2_y\) y \(\tilde{s}^2_{\widehat{y}}\), más efectiva habrá sida la regresión, ya que la regresión habrá heredado mucha variabilidad de los datos \(y_i\), \(i=1,\ldots,n\) y la variabilidad del error, \(SS_E\) será pequeña.
El comentario anterior motiva la definición siguiente del coeficiente de determinación para medir la efectividad de la recta de regresión:
El coeficiente de determinación es una cantidad entre 0 y 1: \(0\leq R^2\leq 1\). Entonces, cuánto más próximo a 1 esté dicho coeficiente, más precisa será la recta de regresión.
El coeficiente de determinación se puede expresar en función de la variabilidad del error de la forma siguiente: \[ R^2=\frac{SS_T-SS_E}{SS_T}=1-\frac{SS_E}{SS_T}=1-\frac{\tilde{s}_e^2}{\tilde{s}_y^2}. \]
Se define el coeficiente de correlación lineal \(r_{xy}\) como \(r_{xy}=\frac{\tilde{s}_{xy}}{\tilde{s}_x\cdot \tilde{s}_y}\). Entonces, el coeficiente de determinación \(R^2\) es el cuadrado del coeficiente de correlación lineal: \(R^2 = r_{xy}^2\).
Veamos la demostración de la última propiedad: \[ \begin{array}{rl} R^2 & \displaystyle =\frac{SS_R}{SS_T}=\frac{\sum\limits_{i=1}^n(b_1x_i+b_0-\overline{y})^2}{(n-1)\tilde{s}_y^2} =\frac{\sum\limits_{i=1}^n\left(\dfrac{\tilde{s}_{xy}}{\tilde{s}_x^2}x_i-\dfrac{\tilde{s}_{xy}}{\tilde{s}_x^2}\overline{x}\right)^2}{(n-1)\tilde{s}_y^2}\\ & \displaystyle =\frac{\dfrac{\tilde{s}_{xy}^2}{\tilde{s}_x^4}\sum\limits_{i=1}^n(x_i-\overline{x})^2}{(n-1)\tilde{s}_y^2} =\dfrac{\tilde{s}_{xy}^2}{\tilde{s}_x^4}\cdot \frac{\tilde{s}_x^2}{\tilde{s}_y^2}=\frac{\tilde{s}_{xy}^2}{\tilde{s}_x^2\cdot \tilde{s}_y^2}=r_{xy}^2 \end{array} \]
Ejemplo
Calculemos las variabilidades anteriores y el coeficiente de determinación para los datos de nuestro ejemplo:
- Variabilidad total:
## [1] 331.3- Variabilidad de la regresión:
## [1] 309.6- Variabilidad del error:
## [1] 21.75Comprobemos que se cumple \(SST=SSR+SSE\):
## [1] 0El coeficiente de determinación \(R^2\) será:
## [1] 0.9344Otra manera de calcularlo es:
## [1] 0.9344En este caso, la regresión explica un 93.44% de la variabilidad de los datos.
R.
Para hallar el coeficiente de determinación en R hemos de usar las funciones lm y summary junto con el parámetro r.squared:
Si aplicamos las funciones anteriores a los datos de nuestro ejemplo, obtenemos:
## [1] 0.9344Usar solamente el coeficiente de determinación para medir la calidad de la regresión es un error.
Tenemos que observar más información para poder afirmar que la regresión obtenida es adecuada y se ajusta bien a nuestros datos.
En R existe una tabla de datos denominada anscombe que pone de manifiesto este hecho.
Vamos a echarle un vistazo:
## 'data.frame': 11 obs. of 8 variables:
## $ x1: num 10 8 13 9 11 14 6 4 12 7 ...
## $ x2: num 10 8 13 9 11 14 6 4 12 7 ...
## $ x3: num 10 8 13 9 11 14 6 4 12 7 ...
## $ x4: num 8 8 8 8 8 8 8 19 8 8 ...
## $ y1: num 8.04 6.95 7.58 8.81 8.33 ...
## $ y2: num 9.14 8.14 8.74 8.77 9.26 8.1 6.13 3.1 9.13 7.26 ...
## $ y3: num 7.46 6.77 12.74 7.11 7.81 ...
## $ y4: num 6.58 5.76 7.71 8.84 8.47 7.04 5.25 12.5 5.56 7.91 ...Vemos que tiene 4 parejas de valores \((x_i,y_i)\) de tamaño 11:
## x1 x2 x3 x4 y1 y2 y3 y4
## 1 10 10 10 8 8.04 9.14 7.46 6.58
## 2 8 8 8 8 6.95 8.14 6.77 5.76
## 3 13 13 13 8 7.58 8.74 12.74 7.71
## 4 9 9 9 8 8.81 8.77 7.11 8.84
## 5 11 11 11 8 8.33 9.26 7.81 8.47
## 6 14 14 14 8 9.96 8.10 8.84 7.04
## 7 6 6 6 8 7.24 6.13 6.08 5.25
## 8 4 4 4 19 4.26 3.10 5.39 12.50
## 9 12 12 12 8 10.84 9.13 8.15 5.56
## 10 7 7 7 8 4.82 7.26 6.42 7.91
## 11 5 5 5 8 5.68 4.74 5.73 6.89Si calculamos los coeficientes de determinación para las 4 parejas, obtenemos un resultado similar:
## [1] 0.6665## [1] 0.6662## [1] 0.6663## [1] 0.6667En cambio, si vemos su representación gráfica, su aspecto es muy distinto:
par(mfrow=c(2,2))
plot(y1~x1,data=anscombe)
abline(lm(y1~x1,data=anscombe),col=2)
plot(y2~x2,data=anscombe)
abline(lm(y2~x2,data=anscombe),col=2)
plot(y3~x3,data=anscombe)
abline(lm(y3~x3,data=anscombe),col=2)
plot(y4~x4,data=anscombe)
abline(lm(y4~x4,data=anscombe),col=2)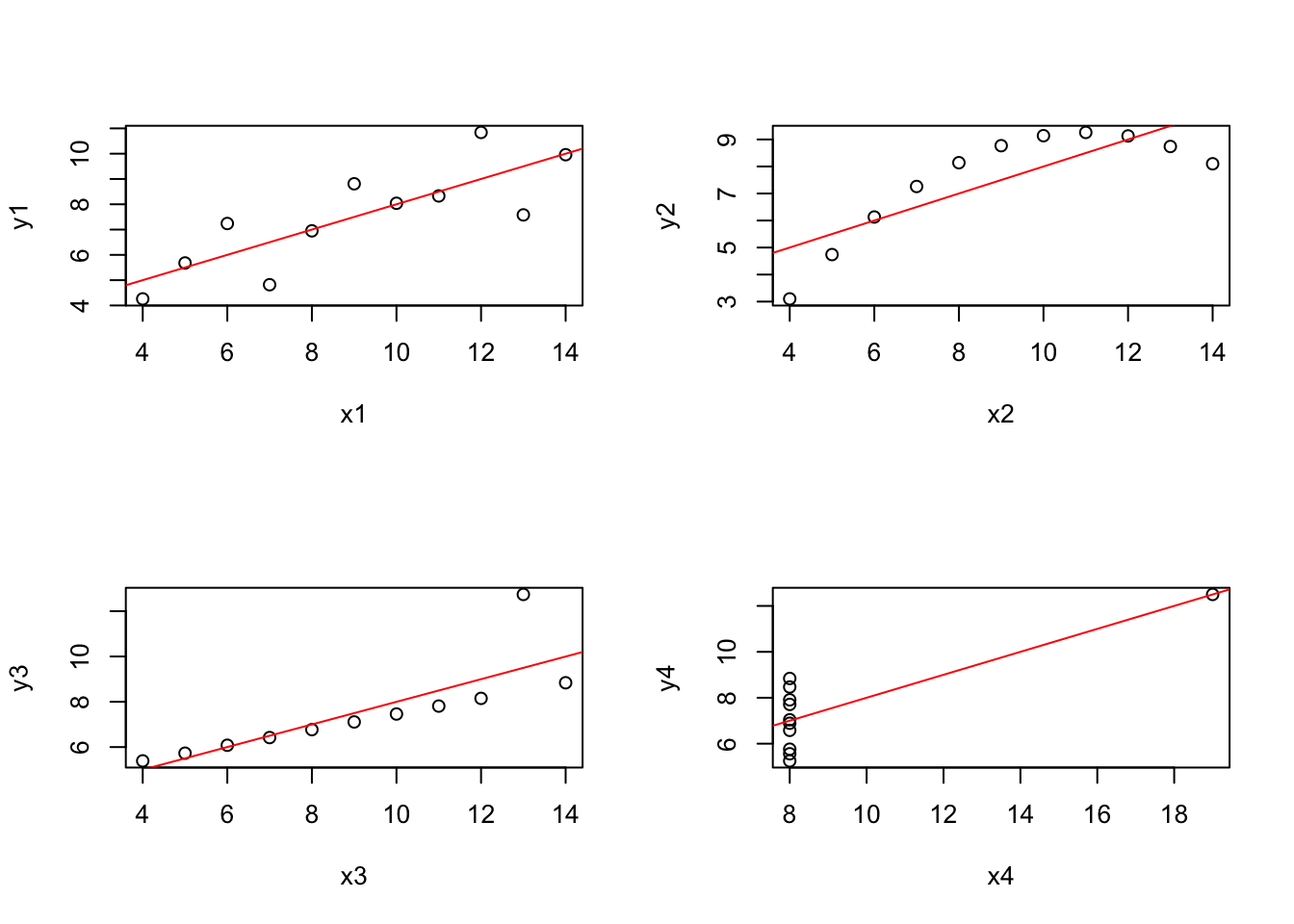
Observamos que en el caso de la tabla de datos (x3,y3), la recta de regresión ha sido efectiva pero en los demás hemos obtenido un error considerable donde el peor caso es el de la tabla de datos (x4,y4).
Por tanto, considerar sólo el valor del coeficiente de determinación para medir el ajuste de la recta de regresión a nuestros datos no es conveniente.
8.1.4 Intervalos de confianza
Para poder hallar los intervalos de confianza al \(100\cdot (1-\alpha)\)% de confianza sobre los parámetros \(\beta_0\) y \(\beta_1\), necesitamos el supuestos siguiente:
Para cada valor \(x_i\) de la variable \(X\), las variables aleatorias \(E_{x_i}\) sigue una distribución normal con media \(\mu_{E_{x_i}}=0\) y varianza \(\sigma_E^2\) constante independiente del valor \(x_i\). También supondremos que dados \(x_i\) y \(x_j\) dos valores distintos de la variable \(X\), la covarianza entre las variables \(E_{x_i}\) y \(E_{x_j}\) es nula: \(\sigma(E_{x_i},E_{x_j})=0\).
Ejemplo
Comprobemos para los datos de nuestro ejemplo si los errores siguen una distribución normal usando el test de Kolmogorov-Smirnov-Lilliefors:
##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: errores
## D = 0.28, p-value = 0.1Como el p-valor es grande, podemos concluir que no tenemos evidencias suficientes para rechazar que los errores siguen una distribución normal.
Bajo las hipótesis anteriores tenemos los dos resultados siguientes:
Entonces bajo el supuesto anterior, los intervalos de confianza para los parámetros \(\beta_1\) y \(\beta_0\) al \(100\cdot (1-\alpha)\)% de confianza son los siguientes:
\(\beta_1\): \(\left] b_1-t_{n-2,1-\frac{\alpha}2} \frac{S}{\tilde{s}_x\sqrt{n-1}}, b_1+t_{n-2,1-\frac{\alpha}2} \frac{S}{\tilde{s}_x\sqrt{n-1}}\right[.\) Lo escribiremos para simplificar de la forma siguiente: \(\beta_1=b_1\pm t_{n-2,1-\frac{\alpha}2} \frac{S}{\tilde{s}_x\sqrt{n-1}}\).
\(\beta_0\): \(b_0\pm t_{n-2,1-\frac{\alpha}2}S\sqrt{\frac{1}{n}+\frac{\overline{x}^2}{(n-1)\tilde{s}_x^2}}\).
Ejemplo
Hallemos los intervalos de confianza para el 95% de confianza para los parámetros \(\beta_1\) y \(\beta_0\) usando los datos del ejemplo que hemos ido desarrollando:
alpha=0.05
S=sqrt(estimación.varianza)
extremo.izquierda.b1 = b1-qt(1-alpha/2,n-2)*S/(sd(sal)*sqrt(n-1))
extremo.derecha.b1 = b1+qt(1-alpha/2,n-2)*S/(sd(sal)*sqrt(n-1))
print('Intervalo confianza para b1:')## [1] "Intervalo confianza para b1:"## [1] 4.004 8.666extremo.izquierda.b0 = b0-qt(1-alpha/2,n-2)*S*sqrt(1/n+media.sal^2/((n-1)*var(sal)))
extremo.derecha.b0 = b0+qt(1-alpha/2,n-2)*S*sqrt(1/n+media.sal^2/((n-1)*var(sal)))
print('Intervalo confianza para b0:')## [1] "Intervalo confianza para b0:"## [1] 77.87 94.87R.
Para hallar los intervalos de confianza de los parámetros \(\beta_1\) y \(\beta_0\) en R hay que aplicar la función confint al objeto lm(...). El parámetro level nos da el nivel de confianza cuyo valor por defecto es 0.95.
Para los datos de nuestro ejemplo, los intervalos de confianza para los parámetros \(\beta_0\) y \(\beta_1\) serían los siguientes en R:
## 2.5 % 97.5 %
## (Intercept) 77.869 94.873
## sal 4.004 8.666Fijado un valor \(x_0\) de la variable \(X\), podemos considerar dos parámetros a estudiar: el valor medio de la variable aleatoria \(Y|x_0\), \(\mu_{Y|x_0}\) y el valor estimado \(y_0\) por la recta de regresión.
Dichos intervalos nos ayudan a estudiar cómo se comporta la regresión cuando el valor de la variable \(X\) vale un determinado valor \(x_0\).
El estimador de los parámetros anteriores, \(\mu_{Y|x_0}\) y \(y_0\) es el mismo: \(\widehat{y}_0 = b_0+b_1\cdot x_0\) pero los errores estándares cambian dependiendo del parámetro que consideremos como indican los dos resultados siguientes:
Bajo el supuesto anterior, sea \(x_0\) un valor concreto de la variable \(X\). Entonces:
Usando el resultado anterior, se tiene que la variable aleatoria: \(\frac{\widehat{y}_0-\mu_{Y/x_0}}{S\sqrt{\frac1{n}+\frac{(x_0-\overline{x})^2}{(n-1) \tilde{s}^2_x}}}\) sigue una ley \(t\) de Student con \(n-2\) grados de libertad.
Usando el resultado anterior, se tiene que la variable aleatoria: \(\frac{\widehat{y}_0-y_0}{S\sqrt{1+\frac1{n}+\frac{(x_0-\overline{x})^2}{(n-1) \tilde{s}^2_x}}}\) sigue una ley \(t\) de Student con \(n-2\) grados de libertad.
A partir de los teoremas anteriores, hallamos los intervalos de confianza para los parámetros \(\mu_{Y|x_0}\) y \(y_0\) al \(100\cdot (1-\alpha)\)% de confianza:
\(\mu_{Y|x_0}\): \(\widehat{y}_0\pm t_{n-2,1-\frac{\alpha}2} S\sqrt{\frac1{n}+\frac{(x_0-\overline{x})^2}{(n-1) \tilde{s}^2_x}}\).
\(y_0\): \(\widehat{y}_0\pm t_{n-2,1-\frac{\alpha}2} S\sqrt{1+\frac1{n}+\frac{(x_0-\overline{x})^2}{(n-1) \tilde{s}^2_x}}\).
Ejemplo
Hallemos un intervalo de confianza para un nivel de sal de \(x_0=4.5\) g. para los parámetros \(\mu_{Y|4.5}\) y \(y_0\) al \(95\)% de confianza:
alpha=0.05
x0=4.5
y0.estimado = b0+b1*x0
extremo.izquierda.mu.x0 = y0.estimado-qt(1-alpha/2,n-2)*S*
sqrt(1/n+(x0-media.sal)^2/((n-1)*var(sal)))
extremo.derecha.mu.x0 = y0.estimado+qt(1-alpha/2,n-2)*S*
sqrt(1/n+(x0-media.sal)^2/((n-1)*var(sal)))
print(paste("Intervalo de confianza para mu de Y para x0=",x0))## [1] "Intervalo de confianza para mu de Y para x0= 4.5"## [1] 111.3 118.5extremo.izquierda.y0 = y0.estimado-qt(1-alpha/2,n-2)*S*
sqrt(1+1/n+(x0-media.sal)^2/((n-1)*var(sal)))
extremo.derecha.y0 = y0.estimado+qt(1-alpha/2,n-2)*S*
sqrt(1+1/n+(x0-media.sal)^2/((n-1)*var(sal)))
print(paste("Intervalo de confianza para y0 para x0=",x0))## [1] "Intervalo de confianza para y0 para x0= 4.5"## [1] 107.5 122.3Para hallar el intervalo de confianza para el parámetros \(\mu_{Y|x_0}\) al \(100\cdot (1-\alpha)\)% de confianza hay que usar la función predict.lm de la forma siguiente:
Para el parámetro \(y_0\) hay que usar la misma función anterior pero cambiando el parámetro interval al valor prediction:
Ejemplo
Hallemos los intervalos de confianza para los parámetros \(\mu_{Y|4.5}\) y \(y_0\) al 95% de confianza usando R:
## fit lwr upr
## 1 114.9 111.3 118.5## fit lwr upr
## 1 114.9 107.5 122.38.1.5 Contraste de hipótesis sobre la pendiente de la recta \(\beta_1\)
Cuando nos planteamos hacer una regresión de la variable \(Y\) sobre la variable \(X\), estamos suponiendo que la variable \(X\) influye en la variable \(Y\). Es decir, que si cambiamos el valor de la variable \(X\), habrá un cambio en la variable \(Y\).
Decir que la variable \(X\) influye en la variable \(Y\) en el contexto de la regresión lineal es equivalente a decir que \(\beta_1\neq 0\) ya que si \(\beta_1=0\), tendríamos que la variación de la variable \(Y\) sólo se debería a fluctuaciones aleatorias.
Por tanto, es interesante plantearse el contraste de hipótesis siguiente sobre el parámetro pendiente de la recta de regresión: \(\beta_1\): \[ \left\{\begin{array}{l} H_0:\beta_1=0,\\ H_1:\beta_1 \neq 0. \end{array} \right. \] En el supuesto de que las variables aleatorias \(E_{x_i}\) son normales \(N(0,\sigma_E^2)\), el estadístico de contraste para realizar el contraste anterior es el siguiente: \(T=\frac{b_1}{\frac{S}{\tilde{s}_x \sqrt{n-1}}},\) que, suponiendo que la hipótesis nula es cierta, se distribuye según una t de Student con \(n-2\) grados de libertad.
Si \(t_0\) es el valor del estadístico de contraste para los valores de nuestra muestra, el p-valor del contraste anterior es el siguiente: \[ p=2\cdot P(t_{n-2}>|t_0|), \] con el significado usual:
- si \(p<0.05\), concluimos que tenemos evidencias suficientes para rechazar la hipótesis nula y, por tanto, tiene sentido la regresión al no rechazar que \(\beta_1\neq 0\),
- si \(p>0.1\), concluimos que no tenemos evidencias suficientes para rechazar la hipótesis nula. En este caso, la regresión no tendría sentido al no rechazar que \(\beta_1=0\) y,
- si \(0.05\leq p\leq 0.1\), estamos en la zona de penumbra. Necesitamos más datos para tomar una decisión clara.
Otra forma de realizar el contraste anterior es observar el intervalo de confianza para \(\beta_1\) y ver si contiene el valor 0.
En caso de contener el valor 0, la regresión no tendría sentido para el nivel de confianza de \(100\cdot (1-\alpha)\)% ya que no rechazamos que \(\beta_1=0\) y en caso de no contener el valor 0, la regresión sí tendría sentido al nivel de confianza anterior.
Ejemplo
Realicemos el contraste anterior para los datos de nuestro ejemplo. El valor del estadístico de contraste será:
## [1] 7.546y el p-valor vale:
## [1] 0.001652Como el p-valor es pequeño, podemos concluir que la regresión tiene sentido en este caso.
Para realizar el contraste anterior en R, hay que estudiar la salida de la función summary aplicada a la función lm:
La salida anterior para los datos de nuestro ejemplo es la siguiente:
##
## Call:
## lm(formula = tensión ~ sal)
##
## Residuals:
## 1 2 3 4 5 6
## 2.23 -2.31 1.46 -1.71 -1.61 1.95
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 86.37 3.06 28.21 0.0000094 ***
## sal 6.33 0.84 7.55 0.0017 **
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 2.33 on 4 degrees of freedom
## Multiple R-squared: 0.934, Adjusted R-squared: 0.918
## F-statistic: 56.9 on 1 and 4 DF, p-value: 0.00165En primer lugar R nos da los errores de los datos cometidos al estimar los valores \(y_i\) por \(\widehat{y}_i\).
A continuación, en una tabla, nos da las estimaciones de los parámetros \(\beta_0\) y \(\beta_1\), \(b_0\) y \(b_1\), en la columna Estimate, los errores estándar de dichos estimadores en la columna Std. Error y el valor del estadístico \(t\) en la columna t value. En la última columna nos da el p-valor para el contraste anterior.
Observamos que hay dos valores de los estadísticos de contraste, uno corresponde al contraste para \(\beta_1\) (la segunda fila) y otro corresponde al contraste para \(\beta_0\) que no tiene ningún interés para ver si la regresión tiene sentido. Pensemos que el hecho de que el parámetro \(\beta_0\) sea nulo no contradice el hecho de que la variable \(X\) tenga efecto en la variable \(Y\).
Por último, en el último párrafo de la salida, nos da el valor del error residual o la estimación de \(S\), el valor del coeficiente de determinación \(R^2\), el Multiple R-squared, y el valor de \(R^2\) ajustado del que hablaremos más adelante.
En la última fila, nos habla del estadístico \(F\) que es otra manera de realizar el contraste sobre el parámetro \(\beta_1\) que hemos explicado anteriormente pero en lugar de usar el estadístico \(T\), se usa el estadístico \(T^2\) que, si la hipótesis nula es cierta, se distribuye según una \(F\) de Fisher con 1 y \(n-2=4\) grados de libertad. Podéis comprobar por ejemplo que \(t_0^2\) vale el valor del F-statistic:
## [1] 56.958.2 Regresión lineal múltiple
En la regresión lineal simple, estudiábamos si una variable dependiente \(Y\) dependía linealmente de una variable independiente o de control \(X\).
En la práctica, dicha situación raramente se da ya que la variable dependiente o de respuesta \(Y\) suele depender de más de una variable de control.
Por tanto, en esta sección vamos a generalizar todo el estudio que hemos hecho para la regresión lineal simple donde sólo hay una variable de control al caso en que tengamos \(k\) variables de control \(X_1,\ldots, X_k\).
En resumen, suponemos que tenemos una variable dependiente \(Y\) y \(k\) variables independientes o de control \(X_1,\ldots, X_k\).
8.2.1 Modelo de regresión lineal múltiple
De forma similar a la regresión lineal simple, suponemos que la relación de la media de la variable aleatoria \(Y\), fijados \(k\) valores de las variables \(X_1,\ldots, X_k\), \(x_1,\ldots,x_k\), es la siguiente: \[ \mu_{Y|x_1,\ldots,x_k}= \beta_0+\beta_1 x_1+\cdots+\beta_k x_k. \] Es decir, la media la media de la variable aleatoria \(Y_{x_1,\ldots,x_k}\) es una función lineal de los valores \(x_1,\ldots,x_k\).
8.2.2 Modelo de regresión lineal múltiple
Los valores \(\beta_0,\beta_1,\ldots,\beta_k\) son los llamados parámetros de regresión y se tienen que estimar a partir de una muestra de las variables consideradas: \[ (x_{i1},x_{i2},\ldots,x_{ik},y_i)_{i=1,\ldots,n} \] Para que dichas estimaciones se puedan realizar, hay que suponer que \(n>k\) ya que en caso contrario tendríamos un problema subestimado: tendríamos más parámetros que valores en la muestra.
Denotaremos el vector \(\underline{x}_i\) al conjunto de los \(k\) valores del individuo \(i\)-ésimo: \(\underline{x}_i= (x_{i1},x_{i2},\ldots,x_{ik})\).
Escribimos el modelo de regresión lineal múltiple de la forma siguiente: \[ Y|x_1,\ldots,x_k=\beta_0+\beta_1 x_1+\cdots+\beta_{k} x_k+E_{x_1,\ldots,x_k}, \] donde
\(Y|x_1,\ldots,x_k\) es la v.a. que da el valor de \(Y\) cuando cada \(X_i\) vale \(x_i\), \(X_i=x_i\),
\(E_{x_1,\ldots,x_k}\) son las v.a. error, o residuales, y representan el error aleatorio del modelo asociado a \((x_1,\ldots,x_k)\).
A partir de una muestra \[ (\underline{x}_{i},y_i)_{i=1,2,\ldots,n} \] vamos a obtener estimaciones \(b_0,b_1,\ldots,b_k\) de los parámetros de regresión \(\beta_0,\beta_1,\ldots,\beta_k\).
Una vez obtenidas las estimaciones \(b_0,b_1,\ldots,b_k\), podemos definir los valores siguientes: \[ \begin{array}{l} \widehat{y}_i=b_0+b_1 x_{i 1}+\cdots+b_{k} x_{i k},\\ y_i=b_0+b_1 x_{i 1}+\cdots+b_{k} x_{i k}+e_i, \end{array} \] donde
\(\widehat{y}_i\) es el valor predicho de a partir de \(y_i\) y \(\underline{x}_{i}\) y
\(e_i\) estima el error \(E_{\underline{x}_{i}}\): \(e_i=y_i-\widehat{y}_i\).
Para simplificar la notación, escribimos los datos de la muestra en forma matricial.
En primer lugar, definimos los vectores siguientes: \[ \mathbf{y}= \left( \begin{array}{l} y_1\\ y_2\\ \vdots\\ y_n \end{array} \right),\ \mathbf{b}=\left( \begin{array}{l} b_0\\ b_1 \\ \vdots\\b_k \end{array} \right),\ \mathbf{\widehat{y}}=\left( \begin{array}{l} \widehat{y}_1\\ \widehat{y}_2\\ \vdots\\\widehat{y}_n \end{array} \right),\ \mathbf{e}=\left( \begin{array}{l} e_1\\ e_2\\ \vdots\\ e_n \end{array} \right). \]
Definimos la matriz \(\mathbf{X}\) a partir de los datos de la muestra de las variables \(X_i\), \(i=1,\ldots,k\): \[ \mathbf{X}=\left( \begin{array}{lllll} 1&x_{11}&x_{12}&\ldots&x_{1k}\\ 1&x_{21}&x_{22}&\ldots&x_{2k}\\ \vdots&\vdots&\vdots&\ddots&\vdots\\ 1&x_{n1}&x_{n2}&\ldots&x_{nk} \end{array} \right). \]
Las ecuaciones \[ \begin{array}{l} \widehat{y}_i=b_0+b_1 x_{i 1}+\cdots+b_{k} x_{i k},\\ y_i=b_0+b_1 x_{i 1}+\cdots+b_{k} x_{i k}+e_i, \end{array} \] se escriben en forma matricial de la forma siguiente: \[ \begin{array}{l} \mathbf{\widehat{y}} = \mathbf{X}\cdot\mathbf{b}, \\ \mathbf{y}= \mathbf{X}\cdot \mathbf{b}+\mathbf{e}.\\ \end{array} \]
8.2.3 Método de los mínimos cuadrados
Definimos el error cuadrático \(SS_E\) cómo: \[ SS_E=\sum\limits_{i=1}^n e^2_i=\sum\limits_{i=1}^n (y_i-\widehat{y}_i)^2 =\sum\limits_{i=1}^n (y_i-b_0-b_1 x_{i 1}-\cdots -b_{k} x_{ik})^2. \]
Los estimadores de los parámetros \(\beta_0,\beta_1,\ldots, \beta_k\) por el método de mínimos cuadrados serán los valores \(b_0,b_1,\ldots, b_k\) que minimicen \(SS_E\).
Para calcularlos, calculamos las derivadas parciales del error cuadrático \(SS_E\) respecto cada \(b_i\), las igualamos a 0, las resolvemos, y comprobamos que la solución \((b_0,\ldots,b_k)\) encontrada corresponde a un mínimo.
El teorema siguiente nos da la expresión de dichos estimadores:
Ejemplo
Se postula que la altura de un bebé (\(y\)) tiene una relación lineal con su edad en días (\(x_1\)), su altura al nacer en cm. (\(x_2\)), su peso en kg. al nacer (\(x_3\)) y el aumento en tanto por ciento de su peso actual respecto de su peso al nacer (\(x_4\))
El modelo es: \[ \mu_{Y|x_1,x_2,x_3,x_4}=\beta_0+\beta_1x_1+\beta_2x_2+\beta_3x_3+\beta_4x_4. \] En una muestra de \(n=9\) niños, se obtuvieron los resultados siguientes:
| \(y\) | \(x_1\) | \(x_2\) | \(x_3\) | \(x_4\) |
|---|---|---|---|---|
| 57.5 | 78 | 48.2 | 2.75 | 29.5 |
| 52.8 | 69 | 45.5 | 2.15 | 26.3 |
| 61.3 | 77 | 46.3 | 4.41 | 32.2 |
| 67 | 88 | 49 | 5.52 | 36.5 |
| 53.5 | 67 | 43 | 3.21 | 27.2 |
| 62.7 | 80 | 48 | 4.32 | 27.7 |
| 56.2 | 74 | 48 | 2.31 | 28.3 |
| 68.5 | 94 | 53 | 4.3 | 30.3 |
| 69.2 | 102 | 58 | 3.71 | 28.7 |
Hallemos los estimadores de los parámetros \(\beta_0,\beta_1,\beta_2,\beta_3\) y \(\beta_4\).
En primer lugar, hallamos la matriz \(\mathbf{X}\) y el vector \(\mathbf{y}\):
\[
\mathbf{X}=\left(
\begin{array}{ccccc}
1&78&48.2&2.75&29.5\\
1&69&45.5&2.15&26.3\\
1&77&46.3&4.41&32.2\\
1&88&49&5.52&36.5\\
1&67&43&3.21&27.2\\
1&80&48&4.32&27.7\\
1&74&48&2.31&28.3\\
1&94&53&4.3&30.3\\
1&102&58&3.71&28.7
\end{array}
\right),\
\mathbf{y}=\left(
\begin{array}{c}
57.5\\ 52.8\\ 61.3\\ 67\\ 53.5\\ 62.7\\ 56.2\\ 68.5\\ 69.2
\end{array}
\right).
\]
El vector de los valores estimados \(\mathbf{b}\) valdrá:
\[
\mathbf{b}=\left(\mathbf{X}^t\cdot \mathbf{X}
\right)^{-1}\cdot \left(\mathbf{X}^t \cdot \mathbf{y}\right).
\]
Realicemos los cálculos anteriores en R:
X=matrix(c(1,78,48.2,2.75,29.5,1,69,45.5,2.15,26.3,
1,77,46.3,4.41,32.2,1,88,49,5.52,36.5,
1,67,43,3.21,27.2,1,80,48,4.32,27.7,
1,74,48,2.31,28.3,1,94,53,4.3,30.3,
1,102,58,3.71,28.7),nrow=9,byrow=TRUE)
y.bebes=cbind(c(57.5,52.8,61.3,67,53.5,62.7,56.2,68.5,69.2))
(estimaciones.b = solve(t(X)%*%X)%*%(t(X)%*%y.bebes))## [,1]
## [1,] 7.14753
## [2,] 0.10009
## [3,] 0.72642
## [4,] 3.07584
## [5,] -0.03004La función lineal de regresión buscada se: \[ \widehat{y}=7.1475+ 0.1001 x_1+0.7264 x_2 +3.0758 x_3 -0.03 x_4. \] Podemos observar, por ejemplo, que cuánto más edad tenga el niño, más altura tiene, cuánto más peso al nacer, más altura tiene pero cuánto más haya aumentado su peso respecto de su peso al nacer, su altura disminuye aunque dicha disminución es minúscula.
8.2.4 Cálculo de la función de regresión en R
Para calcular la función de regresión en R hay que usar la función lm:
En nuestro ejemplo, se calcula de la forma siguiente:
##
## Call:
## lm(formula = y.bebes ~ X[, 2] + X[, 3] + X[, 4] + X[, 5])
##
## Coefficients:
## (Intercept) X[, 2] X[, 3] X[, 4] X[, 5]
## 7.148 0.100 0.726 3.076 -0.030La función de regresión pasa por el vector medio \((\overline{x}_1,\overline{x}_2,\ldots,\overline{x}_k,\overline{y})\): \[ \overline{y}=b_0+b_1 \overline{x}_1+\cdots+b_k \overline{x}_k. \]
La media de los valores estimados se igual a la media de los observados: \[ \overline{\widehat{y}}=\overline{y}. \]
Los errores \((e_i)_{i=1,\ldots,n}\) tienen media 0 y varianza: \[ \tilde{s}_e^2=\frac{SS_E}{n-1}. \]
Verifiquemos las propiedades anteriores para los datos de nuestro ejemplo:
- La función de regresión pasa por el vector medio \((\overline{x}_1,\overline{x}_2,\ldots,\overline{x}_k,\overline{y})\):
## [,1]
## [1,] 0- La media de los valores estimados se igual a la media de los observados:
## [1] 0- Los errores \((e_i)_{i=1,\ldots,n}\) tienen media 0 y varianza \(\tilde{s}_e^2=\frac{SS_E}{n-1}.\)
## [1] 0## [,1]
## [1,] 08.2.5 Coeficiente de determinación
Al igual que hicimos que la regresión lineal simple, vamos a definir el coeficiente de determinación que es una manera (no la única como ya hemos comentado) de medir lo efectiva que es la regresión.
Introducimos las variabilidades siguientes tal como hicimos en la regresión lineal simple:
Variabilidad total o suma total de cuadrados: \(SS_T =\sum\limits_{i=1}^n(y_i-\overline{y})^2 = (n-1)\cdot \tilde{s}_y^2.\)
Variabilidad de la regresión o suma de cuadrados de la regresión: \(SS_R=\sum\limits_{i=1}^n(\widehat{y}_i-\overline{y})^2=(n-1)\cdot \tilde{s}_{\widehat{y}}^2.\)
Variabilidad del error o suma de cuadrados del error: \(SS_E=\sum\limits_{i=1}^n(y_i-\widehat{y}_i)^2=(n-1)\cdot \tilde{s}_e^2.\)
Recordemos que tal como pasaba en la regresión lineal simple, la variabilidad total se puede descomponer como la suma de la variabilidad de la regresión y la variabilidad del error.
Definimos el coeficiente de determinación en una regresión lineal múltiple como: \(R^2=\frac{SS_R}{SS_T}=\frac{\tilde{s}^2_{\widehat{y}}}{\tilde{s}^2_y},\) y representa la fracción de la variabilidad de \(y\) que queda explicada por la variabilidad del modelo de regresión lineal.
De la misma manera, definimos el coeficiente de correlación múltiple de \(y\) respecto de \(x_1,\ldots, x_k\) como \(R=\sqrt{R^2}\).
El coeficiente de determinación verifica las dos primeras propiedades que verificaba el coeficiente de determinación en el caso de la regresión lineal simple:
El coeficiente de determinación es una cantidad entre 0 y 1: \(0\leq R^2\leq 1\). Entonces, cuánto más próximo a 1 esté dicho coeficiente, más precisa será la recta de regresión.
El coeficiente de determinación se puede expresar en función de la variabilidad del error de la forma siguiente: \[ R^2=\frac{SS_T-SS_E}{SS_T}=1-\frac{SS_E}{SS_T}=1-\frac{\tilde{s}_e^2}{\tilde{s}_y^2}. \]
Vamos a calcular las variabilidades y el coeficiente de determinación para los datos del ejemplo que estamos trabajando:
- Variabilidad total:
## [1] 321.2- Variabilidad de la regresión:
## [1] 318.3- Variabilidad del error
## [1] 2.966Comprobemos que la variabilidad total se descompone en la suma de la variabilidad de la regresión y la variabilidad del error:
## [1] 0El coeficiente de determinación será:
## [1] 0.9908o, si se quiere,
## [,1]
## [1,] 0.9908R.
Para hallar el coeficiente de determinación en R podemos usar las mismas funciones que usábamos en la regresión lineal simple:
8.2.6 Coeficiente de determinación ajustado
El coeficiente de determinación definido anteriormente aumenta si aumentamos el número de variables independientes \(k\), incluso si éstas aportan información redundante o poca información. Por ejemplo, si añadimos variables que son linealmente dependientes de las demás.
Para evitar este problema o para penalizar el aumento de variables independientes se usa en su lugar el coeficiente de regresión ajustado: \[ R^2_{adj}=\frac{MS_T-MS_E}{MS_T}, \] donde \(MS_T=\frac{SS_T}{n-1},\quad MS_E=\frac{SS_E}{n-k-1}.\)
Fijarse ahora que si aumentamos \(k\), el valor de \(MS_E\) aumenta y el valor de \(R^2_{adj}\) disminuirá. Por tanto, con el \(R^2_{adj}\) penalizamos el aumento de variables independientes.
Si aumentamos el número de variables independientes con variables explicativas que aporten mucha información a la regresión, el valor de \(SS_E\) disminuirá haciendo que se mantenga estable el valor de \(MS_E\) y el valor de \(R^2_{adj}\) no disminuirá.
La relación entre el coeficiente de determinación ajustado y el coeficiente de determinación es la siguiente: \[ R^2_{adj}=1-(1-R^2)\frac{n-1}{n-k-1}. \]
Calculemos el coeficiente de determinación ajustado para los datos de nuestro ejemplo:
## [,1]
## [1,] 0.9815Observamos que obtenemos un coeficiente de determinación ajustado menor que el coeficiente de determinación.
En general, lo que hemos observado en el ejemplo anterior, ocurre siempre, es decir \(0\leq R^2_{adj} < R^2\leq 1\), lo que nos permite concluir que para el coeficiente de determinación ajustado, es más difícil obtener un valor cercano a 1 que para el coeficiente de determinación.
R.
El cálculo del coeficiente de determinación ajustado en R es parecido al cálculo del coeficiente de determinación: sólo hemos de cambiar el parámetro r.squared por adj.r.squared
8.2.7 Comparación de modelos
Imaginemos que tenemos el modelo \[ Y_{|x_1,\ldots,x_k}=\beta_0 + \beta_1 x_1+\cdots + \beta_k x_k + E_{x_1,\ldots,x_k}, \] y añadimos una nueva variable independiente \(x_{k+1}\). Entonces, tendremos otro modelo: \[ Y_{|x_1,\ldots,x_k,x_{k+1}}=\beta_0 + \beta_1 x_1+\cdots + \beta_k x_k +\beta_{k+1}+ E_{x_1,\ldots,x_k,x_{k+1}}. \] ¿Cómo podemos comparar los dos modelos anteriores?
O, dicho en otras palabras, ¿cómo decidir si la nueva variable introducida \(x_{k+1}\) aporta información relevante?
Una manera de realizar dicha comparación es usando el coeficiente de regresión ajustado \(R^2_{adj}\): el modelo que tenga mayor \(R^2_{adj}\) es el mejor.
Por tanto, si el valor del coeficiente de regresión ajustado del segundo modelo supera el coeficiente de regresión ajustado del primero, diremos que la nueva variable \(x_{k+1}\) aporta información relevante y hay que tenerla en cuenta.
Con los datos de nuestro ejemplo, consideremos sólo la variable independiente correspondiente a la segunda columna de la matriz \(\mathbf{X}\), variable que corresponde a la edad del niño en días.
El coeficiente de correlación ajustado de este modelo que, por cierto, sería de regresión lineal simple vale:
## [1] 0.8823Consideremos ahora las dos primeras variables correspondientes a las columnas segunda y tercera de la matriz \(\mathbf{X}\), variables que corresponden a la edad del niño en días y su altura en nacer.
El coeficiente de correlación ajustado de este modelo vale:
## [1] 0.9617Hemos obtenido un coeficiente de regresión ajustado superior. Por tanto, ha valido la pena añadir la variable correspondiente a la altura al nacer del niño ya que nos ha aportado información relevante al modelo.
Consideremos ahora las tres primeras variables correspondientes a las columnas segunda, tercera y cuarta de la matriz \(\mathbf{X}\), variables que corresponden a la edad del niño en días, su altura en nacer y su peso al nacer.
El coeficiente de correlación ajustado de este modelo vale:
## [1] 0.9851Hemos vuelto a obtener un coeficiente de regresión ajustado superior al anterior. Por tanto, también ha valido la pena añadir la variable peso del niño al nacer al aportar dicha variable información relevante al modelo.
Recordemos que el coeficiente de regresión ajustado del modelo completo era 0.9815, valor inferior al obtenido anteriormente.
Por tanto, la última variable, el aumento del peso actual del niño respecto de su peso al nacer, no aporta información relevante y no debe ser considerada en el modelo de regresión lineal múltiple.
Comparar dos modelos usando el coeficiente de regresión ajustado es uno de los métodos que hay en la literatura para ver si un método es más adecuado que otro.
Existen más métodos como son el AIC (Akaike’s Information Criterion) o el método BIC (Bayesian Information Criterion).
El método AIC cuantifica cuánta información de \(Y\) se pierde con el modelo y cuántas variables usamos: el mejor modelo es el que tiene un valor de AIC más pequeño. Concretamente, se calcula la cantidad siguiente: \(AIC=n\ln(SS_E/n)+2k\) y el modelo con menor AIC es el más adecuado.
Para usar el método AIC en R, hay que usar la función AIC:
Veamos usando el método AIC cuál de los cuatro modelos vistos anteriormente para los datos de nuestro ejemplo es el más adecuado:
## [1] 43.26## [1] 33.76## [1] 25.62## [1] 27.55El método AIC concuerda con el método del coeficiente de determinación ajustado. El mejor modelo es el que incluye las variables correspondientes a las columnas 2, 3 y 4 de la matriz \(\mathbf{X}\) que, recordemos, son las variables la edad del niño en días, su altura en nacer y su peso al nacer.
El método BIC cuantifica cuánta información de \(Y\) se pierde con el modelo y cuántas variables y datos usamos: el mejor modelo es el que tiene un valor de BIC más pequeño. Para saber si un modelo es mejor que otro, hay que calcular la cantidad siguiente: \(BIC=n\ln(SS_E/n)+k\ln(n)\) y, como ya hemos comentado, el modelo con menor BIC es el más adecuado.
Para usar el método BIC en R, hay que usar la función BIC:
Veamos usando el método BIC cuál de los cuatro modelos vistos anteriormente para los datos de nuestro ejemplo es el más adecuado:
## [1] 43.85## [1] 34.55## [1] 26.61## [1] 28.73El método BIC concuerda con los dos métodos usados anteriormente: el método del coeficiente de determinación ajustado y el método AIC. El mejor modelo es el que incluye las variables correspondientes a las columnas 2, 3 y 4 de la matriz \(\mathbf{X}\) que, recordemos, son las variables la edad del niño en días, su altura en nacer y su peso al nacer.
8.2.8 Intervalos de confianza
Vamos a calcular los intervalos de confianza para los \(k+1\) parámetros de modelo \(\beta_0,\beta_1,\ldots,\beta_k\).
Para ello, al igual que hicimos en la regresión lineal simple, supondremos que las variables aleatorias error \(E_i=E_{\underline{x}_{i}}\) son incorreladas, es decir, la covarianza entre un par de ellas cualesquiera es cero y todas normales de media 0 y de misma varianza \(\sigma_E^2\).
Antes de dar los intervalos de confianza, necesitamos conocer las propiedades sobre los estimadores de los parámetros anteriores, es decir, si son insesgados, cómo estimar la varianza común \(\sigma_E^2\) y sus errores estándar.
Dichas propiedades vienen dadas en los teoremas siguientes:
la variable aleatoria \(\frac{\beta_i-b_i}{\sqrt{(S^2\cdot (\mathbf{X}^\top \mathbf{X})^{-1})_{ii}}},\) sigue un ley \(t\) de Student con \(n-k-1\) grados de libertad,
un intervalo de confianza del \((1-\alpha)\cdot 100\%\) de confianza para el parámetro \(\beta_i\) es \(b_i\pm t_{n-k-1,1-\frac{\alpha}2}\cdot \sqrt{(S^2\cdot (\mathbf{X}^\top \mathbf{X})^{-1})_{ii}}.\)
Veamos si los errores de los datos del ejemplo que vamos desarrollando se distribuyen normalmente usando el test de Kolmogorov-Smirnov-Lilliefors:
##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: errores
## D = 0.14, p-value = 0.9Como el p-valor obtenido es muy grande, concluimos que no tenemos evidencias suficientes para rechazar la normalidad de los errores.
La estimación de la varianza común \(S^2\) será:
## [1] 0.7414La estimación de la matriz de covarianzas de los estimadores \(b_0,b_1,b_2,b_3,b_4\) es la siguiente:
## [,1] [,2] [,3] [,4] [,5]
## [1,] 270.919 5.32456 -12.52088 -13.743055 -1.399872
## [2,] 5.325 0.11540 -0.26585 -0.325518 -0.017627
## [3,] -12.521 -0.26585 0.61764 0.742418 0.041580
## [4,] -13.743 -0.32552 0.74242 1.121860 -0.005976
## [5,] -1.400 -0.01763 0.04158 -0.005976 0.027710En la matriz anterior, podemos observar que el estimador con más varianza sería \(b_0\) seguido de \(b_3\) que corresponde a la varianza del peso del niño al nacer.
También podemos observar que entre las variables \(x_2\) (altura del niño al nacer) y \(x_3\) (peso del niño al nacer) existe una gran correlación lineal: 0.7424.
Las estimaciones de los errores estándar de los estimadores \(b_0,b_1,b_2,b_3,b_4\) son las siguientes:
## [1] 16.4596 0.3397 0.7859 1.0592 0.1665Un intervalo de confianza para los estimadores \(b_0,b_1,b_2,b_3,b_4\) al 95% de confianza es el siguiente:
- Intervalo de confianza para \(b_0\) al 95% de confianza:
alpha=0.05
c(estimaciones.b[1]-qt(1-alpha/2,n-k-1)*errores.estandar[1],
estimaciones.b[1]+qt(1-alpha/2,n-k-1)*errores.estandar[1])## [1] -38.55 52.85- Intervalo de confianza para \(b_1\) al 95% de confianza:
c(estimaciones.b[2]-qt(1-alpha/2,n-k-1)*errores.estandar[2],
estimaciones.b[2]+qt(1-alpha/2,n-k-1)*errores.estandar[2])## [1] -0.8431 1.0433- Intervalo de confianza para \(b_2\) al 95% de confianza:
c(estimaciones.b[3]-qt(1-alpha/2,n-k-1)*errores.estandar[3],
estimaciones.b[3]+qt(1-alpha/2,n-k-1)*errores.estandar[3])## [1] -1.456 2.908- Intervalo de confianza para \(b_3\) al 95% de confianza:
c(estimaciones.b[4]-qt(1-alpha/2,n-k-1)*errores.estandar[4],
estimaciones.b[4]+qt(1-alpha/2,n-k-1)*errores.estandar[4])## [1] 0.1351 6.0166- Intervalo de confianza para \(b_4\) al 95% de confianza:
c(estimaciones.b[5]-qt(1-alpha/2,n-k-1)*errores.estandar[5],
estimaciones.b[5]+qt(1-alpha/2,n-k-1)*errores.estandar[5])## [1] -0.4922 0.4321R.
Recordemos que para hallar los intervalos de confianza en R había que usar la función confint aplicado al objeto lm(...). El parámetro level nos da el nivel de confianza con valor por defecto 0.95:
Si aplicamos la función anterior a los datos de nuestro ejemplo, obtenemos los intervalos de confianza para los estimadores \(b_0,b_1,b_2,b_3,b_4\) que ya hemos obtenido antes:
## 2.5 % 97.5 %
## (Intercept) -38.5517 52.8467
## X[, 2] -0.8431 1.0433
## X[, 3] -1.4556 2.9084
## X[, 4] 0.1351 6.0166
## X[, 5] -0.4922 0.4321Tal como hemos hecho en la regresión lineal simple, fijados unos valores concretos de las variables independientes \((x_{10},\ldots,x_{k0})\) podemos considerar dos parámetros más a estudiar: el valor medio de la variable aleatoria \(Y|x_{10},\ldots,x_{k0}\), \(\mu_{Y|x_{10},\ldots,x_{k0}}\) y el valor estimado \(y_0=b_0+b_1\cdot x_{10}+\cdots + b_k\cdot x_{k0}\) por la función de regresión.
Recordemos que los dos intervalos de confianza anteriores nos ayudan a interpretar la regresión cuando el valor de las variables \((X_1,\ldots,X_k)\) valen un determinado valor \((x_{10},\ldots,x_{k0})\).
Para realizar la estimación puntual de los dos parámetros anteriores usaremos el mismo estimador \(\hat{y}_0 =b_0+b_1 x_1+\cdots + b_k x_k\) pero tal como pasaba en la regresión lineal simple, los errores estándar no serán los mismos tal como nos dicen el teorema siguiente.
Bajo las hipótesis anteriores,
el error estándar de \(\widehat{y}_0\) como estimador del parámetro \(\mu_{Y|\underline{x}_0}\) es el siguiente: \[ S\sqrt{\mathbf{x}_0\cdot (\mathbf{X}^\top \cdot\mathbf{X})^{-1}\cdot \mathbf{x}_0^\top}, \]
el error estándar de \(\widehat{y}_0\) como estimador de \(y_0\) es el siguiente: \[ S\sqrt{1+\mathbf{x}_0\cdot (\mathbf{X}^\top \cdot\mathbf{X})^{-1}\cdot \mathbf{x}_0^\top}, \] donde \(\mathbf{x}_0 =(1,\underline{x}_0)=(1,x_{01},\ldots,x_{0k})\).
Los estimadores para hallar los intervalos de confianza para los parámetros \(\mu_{Y|x_{10},\ldots,x_{k0}}\) y el valor estimado \(y_0\) vienen dados por el teorema siguiente:
Por último, bajo las hipótesis anteriores, un intervalo de confianza al \(100\cdot (1-\alpha)\)% de confianza para cada uno de los parámetros \(\mu_{Y|x_{10},\ldots,x_{k0}}\) y el valor estimado \(y_0\) es el siguiente:
Parámetro \(\mu_{Y|x_{10},\ldots,x_{k0}}\): \(\hat{y}_0\pm t_{n-k-1,1-\frac{\alpha}{2}}\cdot S\sqrt{\mathbf{x}_0\cdot (\mathbf{X}^\top \cdot \mathbf{X})^{-1}\cdot \mathbf{x}_0^\top}\).
Parámetro \(y_0\): \(\hat{y}_0\pm t_{n-k-1,1-\frac{\alpha}{2}}\cdot S\sqrt{1+\mathbf{x}_0\cdot (\mathbf{X}^\top \cdot \mathbf{X})^{-1}\cdot \mathbf{x}_0^\top}\).
Hallemos un intervalo de confianza para los datos de nuestro ejemplo suponiendo un niño con \(x_{10}=75\) días de edad, de altura \(x_{20}=50\) cm. al nacer, de \(x_{30}=4\) kg. al nacer y con un \(x_{40}=30\)% de aumento de peso con respecto su peso al nacer.
El intervalo de confianza para el parámetro \(\mu_{Y|x_{10}=75,x_{20}=50,x_{30}=4,x_{40}=30}\) al 95% de confianza es el siguiente:
alpha=0.05
x0 = c(1,75,50,4,30)
y0.estimado = sum(estimaciones.b*x0)
c(y0.estimado-qt(1-alpha/2,n-k-1)*sqrt(S2*(t(x0)%*%solve(t(X)%*%X)%*%x0)),
y0.estimado+qt(1-alpha/2,n-k-1)*sqrt(S2*(t(x0)%*%solve(t(X)%*%X)%*%x0)))## [1] 52.99 71.77El intervalo de confianza para el parámetro \(y_0\) al 95% de confianza es el siguiente:
c(y0.estimado-qt(1-alpha/2,n-k-1)*sqrt(S2*(1+(t(x0)%*%solve(t(X)%*%X)%*%x0))),
y0.estimado+qt(1-alpha/2,n-k-1)*sqrt(S2*(1+(t(x0)%*%solve(t(X)%*%X)%*%x0))))## [1] 52.69 72.07R.
Para hallar el intervalo de confianza para el parámetros \(\mu_{Y|x_{10},\ldots,x_{k0}}\) al \(100\cdot (1-\alpha)\)% de confianza hay que usar la función predict.lm de la forma siguiente:
newdata=data.frame(x1=x10,...,xk=xk0)
predict.lm(lm(y~x1+...+xk),newdata,interval="confidence",level= nivel.confianza)Para el parámetro \(y_0\) hay que usar la misma función anterior pero cambiando el parámetro interval al valor prediction:
newdata=data.frame(x1=x10,...,xk=xk0)
predict.lm(lm(y~x1+...+xk),newdata,interval="prediction",level= nivel.confianza)Para los datos del ejemplo anterior, los intervalos de confianza para los parámetros \(\mu_{Y|x_{10}=75,x_{20}=50,x_{30}=4,x_{40}=30}\) y \(y_0\) al 95% de confianza se calcularían en R de la forma siguiente:
newdata=data.frame(x1=75,x2=50,x3=4,x4=30)
x1=X[,2]
x2=X[,3]
x3=X[,4]
x4=X[,5]
predict.lm(lm(y.bebes~x1+x2+x3+x4),newdata,interval="confidence",level= 0.95)## fit lwr upr
## 1 62.38 52.99 71.77## fit lwr upr
## 1 62.38 52.69 72.078.2.9 Contrastes de hipótesis sobre los parámetros \(\beta_i\)
En el caso de la regresión lineal múltiple podemos plantearnos el contraste de hipótesis siguiente: \[ \left.\begin{array}{l} H_0: \beta_1=\beta_2=\cdots=\beta_k=0, \\ H_1: \mbox{existe algún $i$ tal que }\beta_i \neq 0. \end{array} \right\} \] Es decir, queremos contrastar si la regresión lineal múltiple realizada ha tenido sentido ya que la hipótesis nula equivaldría a decir que ninguna variable independiente \(X_i\), \(i=1,\ldots,k\) ha tenido efecto sobre la variable \(Y\) y como consecuencia, la regresión no ha tenido sentido.
Para realizar el contraste de hipótesis anterior, podríamos plantear los \(k\) contrastes siguientes: \[ \left.\begin{array}{l} H_0: \beta_i=0, \\ H_1: \beta_i \neq 0, \end{array} \right\} \] usando como estadístico de contraste \(\frac{\beta_i-b_i}{\sqrt{(S^2\cdot (\mathbf{X}^\top \mathbf{X})^{-1})_{ii}}}\), que sabemos que sigue una ley \(t\) de Student con \(n-k-1\) grados de libertad.
El problema es que los \(k\) contrastes anteriores no son independientes, es decir, los estadísticos de contraste son variables aleatorias no independientes entre otras cosas porque su matriz de covarianzas no tiene por qué ser diagonal.
Por tanto, si realizamos el contraste de hipótesis original a partir de los \(k\) contrastes anteriores, es muy complicado hallar el error tipo I \(\alpha\) del contraste original a partir de los errores tipo I de cada uno de los \(k\) contrastes al fallar la independencia de los mismos.
Otra posibilidad para evitar el problema comentado anteriormente es plantear el contraste original como un contraste ANOVA donde las subpoblaciones consideradas serían las variables \(Y|\underline{x}_1, \ldots, Y|\underline{x}_n\) siendo \(\underline{x}_1, \ldots, \underline{x}_n\), \(n\) valores concretos de las variables independientes \((X_1,\ldots, X_k)\).
Fijarse que si la hipótesis nula \(H_0\) es cierta, es decir \(\beta_1=\cdots =\beta_k=0\), entonces los valores medios de las variables anteriores serían los mismos ya que recordemos que suponemos que: \[ \mu_{Y|\underline{x}}=\beta_0 +\beta_1 \cdot x_1 +\cdots +\beta_k\cdot x_k=\beta_0, \] para todo valor de \(\underline{x}\).
Por tanto, el contraste original sería equivalente a realizar el contraste ANOVA siguiente: \[ \left.\begin{array}{l} H_0:\mu_{Y|\underline{x}_1}=\cdots=\mu_{Y|\underline{x}_n},\\ H_1:\mbox{existen $i$ y $j$ tales que }\mu_{Y|\underline{x}_i}\neq \mu_{Y|\underline{x}_j}. \end{array} \right\} \] Para que el modelo de regresión lineal múltiple tenga sentido hemos de rechazar la hipótesis nula anterior.
La tabla ANOVA es la siguiente:
| Origen variación | Grados de libertad | Sumas de cuadrados | Cuadrados medios | Estadístico de contraste | p-valor |
|---|---|---|---|---|---|
| Regresión | \(k\) | \(SS_R\) | \(MS_R=\frac{SS_R}{k}\) | \(F=\frac{MS_R}{MS_E}\) | p-valor |
| Error | \(n-k-1\) | \(SS_E\) | \(MS_E=\frac{SS_E}{n-k-1}\) |
donde el estadístico de contraste \(F=\frac{MS_R}{MS_E}\), suponiendo la hipótesis nula cierta, sigue la distribución \(F\) de \(k\) y \(n-k-1\) grados de libertad.
El p-valor del contraste anterior vale \(p=P(F_{k,n-k-1}\geq f_0),\) siendo \(f_0\) el valor del estadístico de contraste \(F\) para nuestros datos.
R.
Para calcular la tabla ANOVA en R de una regresión lineal múltiple hay que usar la función anova de la forma siguiente:
donde Xd es una matriz cuyas columnas son los valores de las variables independientes \(x_1,\ldots, x_k\).
Para hallar la tabla ANOVA para los datos de nuestro ejemplo, hemos de hacer lo siguiente:
## Analysis of Variance Table
##
## Response: y.bebes
## Df Sum Sq Mean Sq F value Pr(>F)
## X[, 2:5] 4 318 79.6 107 0.00025 ***
## Residuals 4 3 0.7
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Observamos que el p-valor es muy pequeño. Por tanto, rechazamos la hipótesis nula y concluimos que la regresión ha tenido sentido en este caso.
Otro tipo de contrastes que podemos plantearnos sobre los parámetros del modelo \(\beta_i\) es, si fijado \(i\), \(\beta_i\) aporta algo al modelo, o, si la variable \(x_i\) es significativa.
Concretamente, fijado \(i\), nos planteamos el contraste siguiente: \[ \left.\begin{array}{l} H_0: \beta_i=0, \\ H_1: \beta_i \neq 0, \end{array} \right\} \] usando como estadístico de contraste \(\frac{\beta_i-b_i}{\sqrt{(S^2\cdot (\mathbf{X}^\top \mathbf{X})^{-1})_{ii}}}\), que sabemos que sigue una ley \(t\) de Student con \(n-k-1\) grados de libertad.
El p-valor del contraste anterior vale \(p=2\cdot P(t_{n-k-1}>|t_0\), donde \(t_0\) es el valor obtenido por el estadístico de contraste usando nuestros datos.
Para que la variable \(x_i\) sea significativa o para que aporte información relevante al modelo de regresión lineal múltiple, debemos rechazar la hipótesis nula en el contraste anterior u obtener un p-valor pequeño.
R.
Para realizar todos los contrastes anteriores en R hay que usar la función summary aplicada al objecto lm(...):
y R nos da toda la información sobre la regresión múltiple realizada.
Si aplicamos la función summary a los datos de nuestro ejemplo, obtenemos la salida siguiente:
##
## Call:
## lm(formula = y.bebes ~ x1 + x2 + x3 + x4)
##
## Residuals:
## 1 2 3 4 5 6 7 8 9
## -0.0405 -0.1290 0.2150 -0.4324 -0.6461 0.2214 0.5225 1.1276 -0.8385
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 7.147 16.460 0.43 0.687
## x1 0.100 0.340 0.29 0.783
## x2 0.726 0.786 0.92 0.408
## x3 3.076 1.059 2.90 0.044 *
## x4 -0.030 0.167 -0.18 0.866
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.861 on 4 degrees of freedom
## Multiple R-squared: 0.991, Adjusted R-squared: 0.982
## F-statistic: 107 on 4 and 4 DF, p-value: 0.000254En primer lugar R nos da la lista de los errores cometidos en las estimaciones.
En segundo lugar, R nos muestra una tabla con las columnas siguientes:
Estimate: los valores estimados de los parámetros \(\beta_0,\beta_1,\beta_2,\beta_3\) y \(\beta_4\). Es decir, los valores \(b_0,b_1,b_2,b_3\) y \(b_4\).Std. Error: los errores estándares de los estimadores \(b_0,b_1,b_2,b_3\) y \(b_4\).t value: el valor del estadístico de contraste cuando realizamos el contraste \(\left.\begin{array}{l} H_0: \beta_i=0, \\ H_1: \beta_i \neq 0, \end{array} \right\}\) sobre cada parámetro \(\beta_i\), \(i=0,1,2,3,4\).Pr(>|t|): los p-valores del contraste anterior.
Observamos que todas las variables excepto la variable x3, peso del niño al nacer, son no significativas para el modelo.
A continuación nos da el error residual que es la estimación de \(\sqrt{S^2}\), el valor de coeficiente de determinación \(R^2\) y el coeficiente de determinación ajustado \(R^2_{adj}\).
Para finalizar nos da el valor del estadístico de contraste ANOVA comentado anteriormente junto con el p-valor del mismo.
8.2.10 Diagnósticos. Estudio de los residuos
Para que el modelo de regresión lineal tanto simple como múltiple sea fiable en las conclusiones derivadas de las estimaciones e inferencias (intervalos de confianzas, contrastes de hipótesis) que realizamos a partir de dicho modelo, se tienen que verificar unas hipótesis.
Las tareas que realizan dichas verificaciones se denominan diagnósticos de regresión.
Los diagnósticos de regresión se clasifican en tres categorías:
Errores: los errores tienen que seguir una \(N(0,\sigma)\), con la misma varianza, y ser incorrelados.
Modelo: los puntos se tienen que ajustar a la estructura lineal considerada.
Observaciones anómalas: a veces unas cuántas observaciones no se ajustan al modelo y hay que detectarlas.
8.2.11 Tipos de diagnósticos de regresión
Hay dos métodos usados en los diagnósticos de regresión:
- Métodos gráficos: son métodos muy flexibles pero difíciles de interpretar.
- Métodos numéricos: son métodos de utilidad más limitada con respecto a los métodos gráficos pero con interpretación inmediata.
8.2.12 Distribución de los errores
Uno de los problemas que puede sufrir nuestro modelo es que la varianza de los residuos no sea constante. Veamos uno ejemplo
set.seed(2020)
x<-runif(100)
y<-1-2*x+0.3*x*rnorm(100)
par(mfrow=c(1,2))
plot(x,y)
rlm=lm(y~x)
abline(rlm,col="red")
plot(rlm$res~rlm$fitted.values,xlab="Valores ajustados",ylab="Residuos del modelo")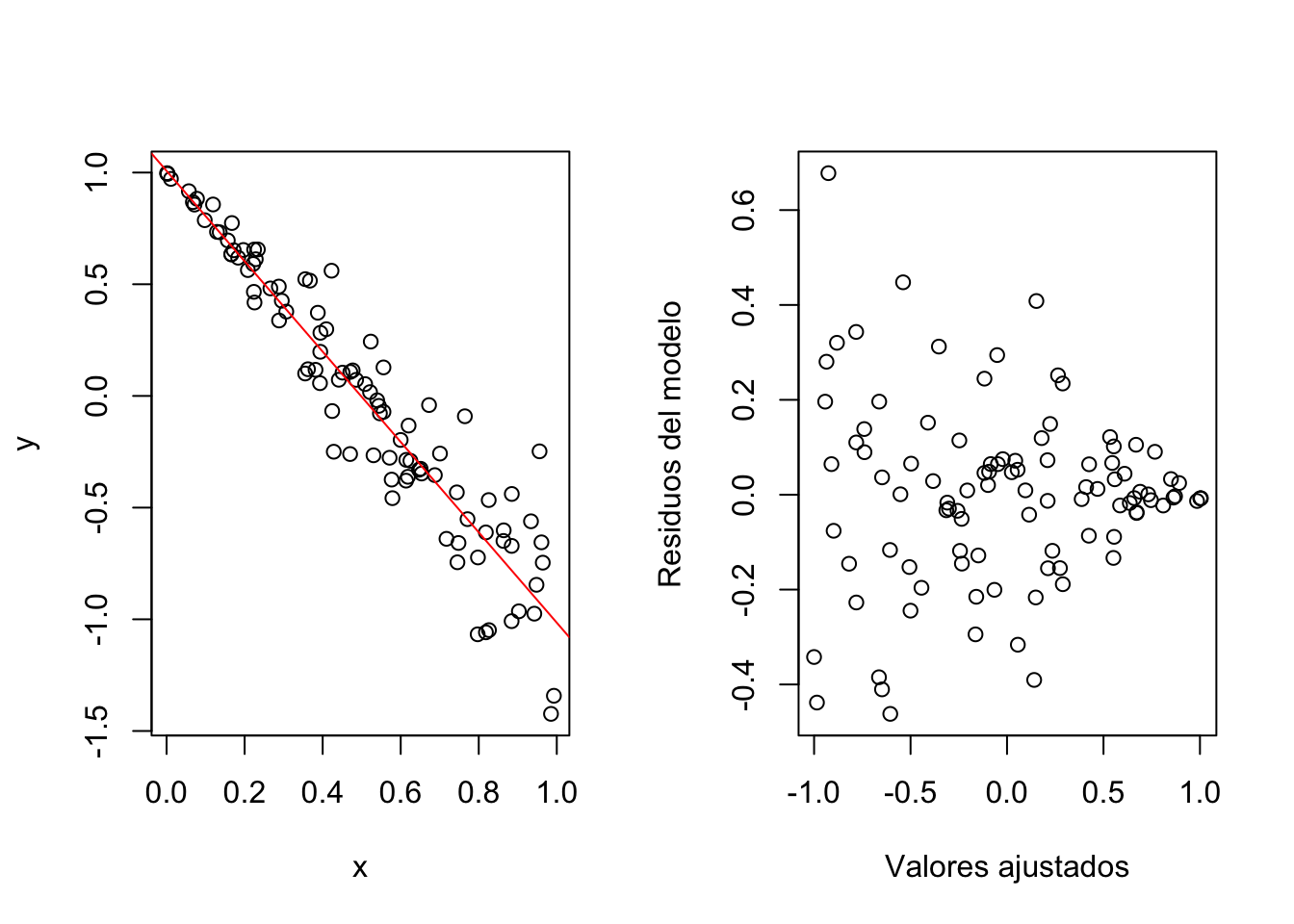
Como podemos observar, hemos realizado una regresión simple de 100 puntos cuyas coordenadas \(x\) son valores aleatorios distribuidos uniformemente en el intervalo \((0,1)\) y cuyas coordenadas \(y\) son valores ajustados a la recta \(y=1-2x\) añadiendo un “ruido” normal estándar amortiguado con un coeficiente de 0.3.
El gráfico de la izquierda muestra los 100 puntos junto con la correspondiente recta de regresión en color rojo.
El gráfico de la derecha muestra la distribución de los pares de los errores del modelo vs. los valores ajustados, \((\hat{y}_i,e_i)\), \(i=1,\ldots,n=100\). Si el modelo fuese homocedástico, es decir, que la varianza de los residuos fuese la misma para cualquier valor \(x\), observaríamos una distribución de puntos uniforme, o lo que coloquialmente se llama un “cielo estrellado”.
En cambio, observamos una distribución “triangular” o “en forma de cuña” donde a medida que aumenta el valor ajustado de los puntos, disminuye la dispersión o la variación de los errores, hecho que nos detecta que dicho modelo es anómalo en el sentido de no ser homocedástico.
El método gráfico anterior, es decir, el gráfico de los errores \(e_i\) en función de los valores estimados \(\hat{y}_i\) es un método para “observar” gráficamente si el modelo es homocedástico.
Existe un método numérico para detectar la homocedasticidad del modelo llamado Test de White.
8.2.13 Test de White
Para aplicar el Test de White, hay que seguir los pasos siguientes:
Obtener los \(\{e_i\}_{i=1,\ldots,n}\) residuos de la regresión lineal inicial.
Calcular el coeficiente de determinación \(R^2\) de la regresión lineal de los \(e_i^2\) respecto de las variables iniciales, sus cuadrados y los productos cruzados dos a dos. Es decir, calcular el coeficiente de determinación del modelo de regresión siguiente: \[ \begin{array}{rl} \mu_{E^2|x_1,\ldots,x_k,x_1^2, \ldots,x_k^2,x_ix_j(i,j=1,\ldots,n,i<j)} = & \beta_{0}+\beta_{1} x_1+\cdots +\beta_{k} x_k+\\ & \beta_{1}^{(2)} x_1^2+\cdots + \beta_k^{(2)}x_k^2+\\ & \beta_{12} x_1 x_2+\cdots + \beta_{k-1,k}x_{k-1} x_k. \end{array} \]
Calcular el estadístico \(X_0=nR^2\), el cual, suponiendo que la varianza es constante, sigue una \(\chi^2_q\), donde \(q\) es el número de variables independientes de la regresión del paso anterior.
Calculamos el p-valor \(P(\chi_q^2\geq X_0)\) con el significado usual.
Ejemplo
Apliquemos el Test de White a los datos del ejemplo anterior:
## [1] 21.17## [1] 0.00002536Obtenemos un valor muy pequeño. Concluimos consecuentemente que tenemos evidencias suficientes para rechazar que las varianzas de los residuos no son iguales para cualquier valor de \(x\) o, dicho en otras palabras, el modelo no es homocedástico, es heterocedástico.
R.
Para usar el Test de White en R, tenemos que usar la función bptest del paquete lmtest.
donde lm1 es el objeto de R donde hemos guardado la información de la regresión original y X es la matriz que contiene los valores de la muestra de las variables independientes.
Si realizamos el Test de White para los datos del ejemplo anterior en R obtenemos lo siguiente:
Ejemplo de los bebés
Recordemos que en este ejemplo teníamos \(k=4\) variables independientes:
- \(x_1\): la edad del bebé en días.
- \(x_2\): su altura al nacer.
- \(x_3\): su peso al nacer.
- \(x_4\): su aumento en tanto por ciento de su peso actual respecto de su peso al nacer.
La tabla de datos constaba de \(n=9\) bebés.
En este caso, realizar el test de White no tendría sentido ya que tendríamos más variables independientes que valores en la muestra en la regresión auxiliar de \(e_i^2\).
Las variables independientes en dichas regresión auxiliar tal como hemos explicado serían: \(x_1,x_2,x_3,x_4,x_1^2,x_2^2,x_3^2,x_4^2,x_1\cdot x_2,x_1\cdot x_3,x_1\cdot x_4,x_2\cdot x_3,x_2\cdot x_4,x_3\cdot x_4\).
Por tanto, tendríamos 14 variables independientes pero sólo \(n=9\) valores de \(e_i^2\). Se violaría la hipótesis en la regresión lineal múltiple de que el número de variables independientes debe ser menor que el número de valores en la muestra.
8.2.14 Test de Breusch-Pagan
Cuando nos encontramos en una situación como la del ejemplo de los bebés, en lugar de aplicar el Test de White para contrastar la homocedasticidad de los residuos, podemos aplicar el Test de Breuch-Pagan.
Su aplicación es bastante parecida al Test de White pero evita los términos de segundo orden en la regresión auxiliar.
Para aplicar el Test de Breusch-Pagan, hay que seguir los pasos siguientes:
Obtener los \(\{e_i\}_{i=1,\ldots,n}\) residuos de la regresión lineal inicial.
Calcular el coeficiente de determinación \(R^2\) de la regresión lineal de los \(e_i^2\) respecto de las variables iniciales. Es decir, calcular el coeficiente de determinación del modelo de regresión siguiente: \[ \mu_{E^2|x_1,\ldots,x_k} = \beta_{0}+\beta_{1} x_1+\cdots +\beta_{k} x_k. \]
Calcular el estadístico \(X_0=nR^2\), el cual, suponiendo que la varianza es constante, sigue una \(\chi^2_k\).
Calculamos el p-valor \(P(\chi_k^2\geq X_0)\) con el significado usual.
Ejemplo de los bebés
Apliquemos el Test de Breusch-Pagan al ejemplo de los bebés.
Los pasos a seguir son:
- Los residuos \(e_i\), \(i=1,\ldots,n=9\) ya se habían calculado anteriormente
## [,1]
## [1,] -0.04053
## [2,] -0.12898
## [3,] 0.21498
## [4,] -0.43238
## [5,] -0.64610
## [6,] 0.22143
## [7,] 0.52245
## [8,] 1.12764
## [9,] -0.83852- Calculamos el coeficiente de determinación del modelo de regresión auxiliar:
## [1] 0.6176- Calculamos el valor del estadístico de contraste \(X_0=nR^2\) y el p-valor correspondiente:
## [1] 5.558## [1] 0.2347Hemos obtenido un p-valor bastante grande. Concluimos consecuentemente que no tenemos indicios suficientes para rechazar la homocedasticidad de los errores en este caso.
R.
Para usar el Test de Breusch-Pagan en R, tenemos que usar la misma función bptest del paquete lmtest.
donde lm1 es el objeto de R donde hemos guardado la información de la regresión original.
Ejemplo de los bebés
Si aplicamos el Test de Breusch-Pagan en R en este ejemplo obtenemos:
y.bebes=c(57.5,52.8,61.3,67,53.5,62.7,56.2,68.5,69.2)
reg.mul.original = lm(y.bebes~X[,2]+X[,3]+X[,4]+X[,5])
bptest(reg.mul.original)##
## studentized Breusch-Pagan test
##
## data: reg.mul.original
## BP = 5.6, df = 4, p-value = 0.2obteniendo los mismos resultados que hemos hallado anteriormente realizando los cálculos “a mano”.
8.2.15 Normalidad de los residuos
Para detectar la normalidad de los residuos, podemos aplicar todos los tests de normalidad vistos en el tema de Bondad de Ajuste:
- Test de Kolmogorov-Smirnov. Usar como parámetros la media y de la desviación típica los valores 0 y el valor de la estimación de \(\sigma\): \(\sqrt{S^2}\).
- Test de Kolmogorov-Smirnov-Lilliefors.
- Test de normalidad de Anderson-Darling.
- Test de Shapiro-Wilks (S-W).
- Test omnibus de D’Agostino-Pearson.
Es aconsejable también realizar las pruebas gráficas de normalidad vistos también en el tema de Bondad de ajuste como los Q-Q-plots.
Ejemplo de los bebés
Realicemos un Q-Q-plot de los residuos en el ejemplo considerado:
library(car)
estimación.sigma2 = sum(errores^2)/(n-k-1)
qqPlot(errores,distribution = "norm", mean=0,sd=sqrt(estimación.sigma2))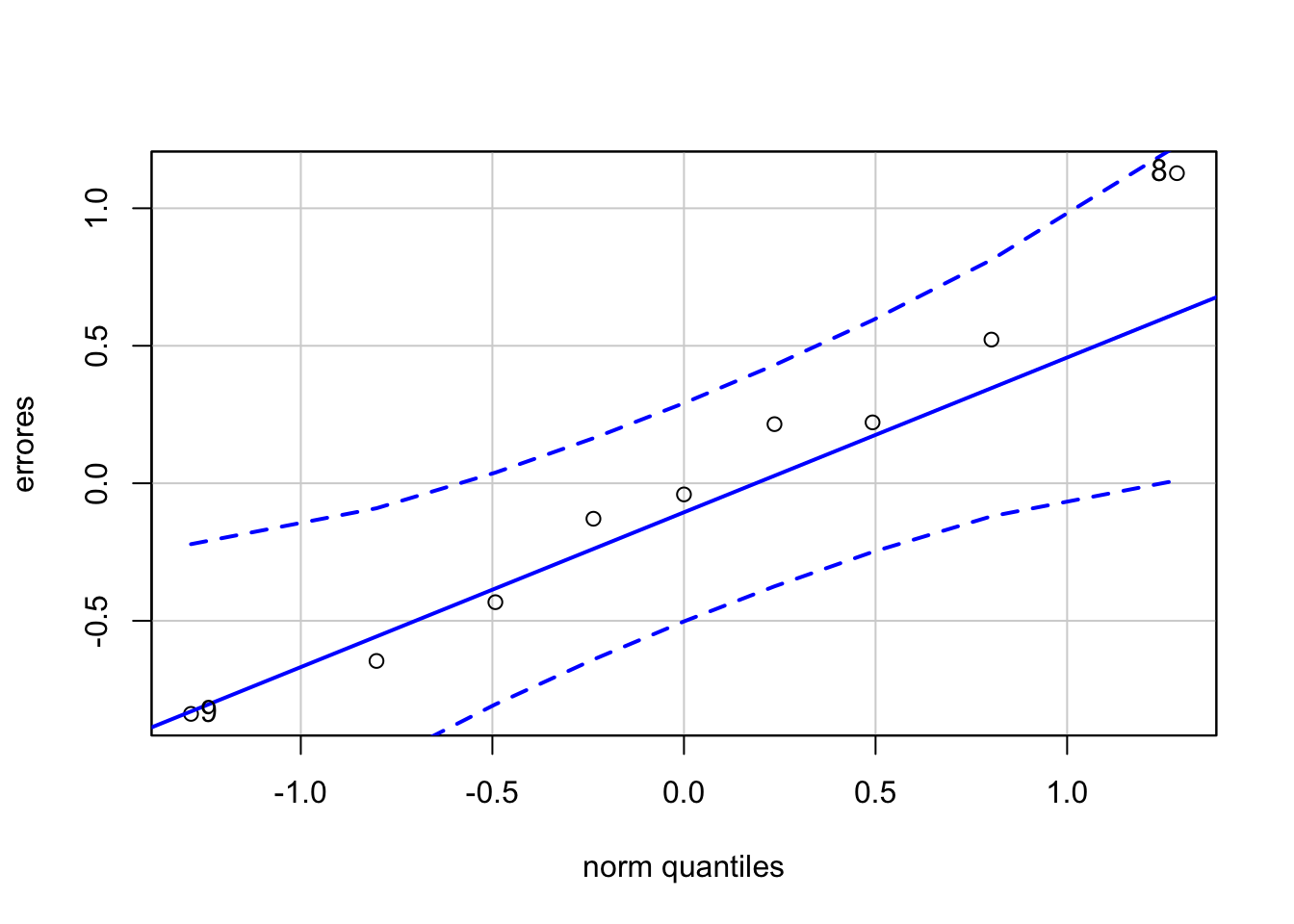
## [1] 8 9Vemos que los \(n=9\) valores de la muestra están dentro de las bandas de confianza, lo que nos permite concluir que gráficamente se observa que los residuos parece que siguen la normalidad.
Recordemos que ya hemos aplicado el test de Kolmogorov-Smirnov-Lilliefors a los residuos, obteniendo:
##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: errores
## D = 0.14, p-value = 0.9un p-valor muy grande, lo que nos permite concluir también que no tenemos indicios suficientes para rechazar la normalidad de los residuos en este caso.
8.2.16 Correlación de los residuos: Test de Durbin-Watson
Otra de las hipótesis que se deben verificar para que el análisis de regresión sea correcto es la incorrelación de los residuos.
La autocorrelación de los residuos puede ser de dos tipos:
- Autocorrelación positiva: un valor positivo (negativo) de un error genera una sucesión de residuos positivos (negativos).
- Autocorrelación negativa: los residuos van alternando de signo.
Para comprobar si se satisface que los residuos no presenten correlación, se puede aplicar el Test de Durbin-Watson.
Expliquemos cómo aplicar dicho test.
Sean \(\{e_i\}_{i=1,\ldots,n}\) los residuos de la regresión.
Sean \(E_i\) y las \(E_{i-1}\) variables aleatorias error (trasladadas en un índice) y la recta de regresión de \(E_i\) con respecto a \(E_{i-1}\): \(E_i=\beta_1E_{i-1}+\beta_0\).
Se plantea el siguiente contraste: \[\left.\begin{array}{ll} H_0: \beta_1=0,\\H_1: \beta_1\neq0,\end{array}\right\}\] con el siguiente estadístico de contraste: \(d=\frac{\sum\limits^n_{i=2}{(e_i-e_{i-1})^2}}{\sum\limits_{i=1}^n{e_i^2}}.\)
El valor de este estadístico es aproximadamente \(2(1-b_1)\) donde \(b_1\) se una estimación de \(\beta_1\).
Si la hipótesis nula \(H_0\) es cierta, su distribución es la de una cierta combinación lineal de \(\chi^2\).
El test necesita de una tabla de valores críticos para tomar la decisión final.
Dicha tabla puede encontrarse en Images Google y escribiendo test de Durbin Watson en la casilla de búsqueda.
Concretamente, \(d\) se tiene que comparar con dos valoras críticos \(d_{L,\alpha}\) y \(d_{U,\alpha}\), donde \(\alpha\) es el nivel de significación que depende de \(n\) y de \(k\).
Decidimos si hay autocorrelación positiva:
- Si \(d<d_{L,\alpha}\), hay autocorrelación positiva.
- Si \(d>d_{U,\alpha}\), no hay autocorrelación positiva.
- De lo contrario, nos encontramos en la zona de penumbra.
Decidimos si hay autocorrelación negativa:
- Si \(4-d<d_{L,\alpha}\), hay autocorrelación negativa.
- Si \(4-d>d_{U,\alpha}\), no hay autocorrelación negativa.
- De lo contrario, nos encontramos en la zona de penumbra.
Ejemplo de los bebés
Vamos a ver si hay autocorrelación para la tabla de datos de los bebés.
El valor del estadístico de contraste \(d\) valdrá en este caso:
## [1] 1.911Si miramos los valores críticos para \(\alpha=0.05\), \(n=9\) y \(k=4\) en la Tabla del estadístico de Durbin-Watson, obtenemos los valores siguientes: \(d_{L,0.05}=0.3\) y \(d_{U,0.05}=2.59\).
A continuación, miramos si hay autocorrelación positiva: como \(1.9106\) está entre \(d_{L,0.05}\) y \(d_{U,0.05}\), estamos en la zona de penumbra y no podemos tomar una decisión clara.
Finalmente, testeamos si hay autocorrelación negativa. El valor de \(4-d\) será: 2.0894. Observamos también que \(4-d\) está entre \(d_{L,0.05}\) y \(d_{U,0.05}\), por tanto, volvemos a estar estamos en la zona de penumbra y no podemos tomar una decisión clara.
En resumen, en este ejemplo no podemos decidir de forma clara a partir del estadístico de Durbin-Watson si los errores están incorrelados .
R.
El test de Durbin-Watson está implementado en la función dwtest del paquete lmtest.
El parámetro alternative nos indica si se testea que la autocorrelación es positiva (greater) o negativa (less):
donde en r se ha guardado el objecto de la regresión lm(y~x1+...+xk) y, como hemos indicado, con alternative=greater testeamos si los residuos tienen autocorrelación positiva y con alternative=less, si tienen autocorrelación negativa.
Ejemplo de los bebés
Para testear si los errores tienen autocorrelación positiva, hacemos lo siguiente:
##
## Durbin-Watson test
##
## data: reg.mul.original
## DW = 1.9, p-value = 0.3
## alternative hypothesis: true autocorrelation is greater than 0Observamos que obtenemos el mismo valor del estadístico de contraste que en los cálculos realizados “a mano”.
Sin embargo, R nos da un p-valor del que podemos concluir que no hay autocorrelación positiva en los residuos. Recordemos que en los cálculos realizados “a mano”, no podíamos tomar una decisión clara.
Esto es debido a que R es capaz de hallar los p-valores a partir de la matriz de los datos \(\mathbf{X}\). Es decir, R usa información de nuestra muestra para hallar los p-valores. Por tanto, si os encontráis una discrepancia de este tipo, la conclusión que os dé R prevalece sobre los cálculos realizados a partir de la tabla del Test de Durbin-Watson.
Si testeamos si los errores tienen autocorrelación negativa, obtenemos:
##
## Durbin-Watson test
##
## data: reg.mul.original
## DW = 1.9, p-value = 0.7
## alternative hypothesis: true autocorrelation is less than 0Obtenemos un p-valor grande, concluyendo a partir de R que no tenemos indicios suficientes para rechazar que los errores no tengan autocorrelación negativa. Dicha conclusión prevalecería sobre la conclusión que hemos realizado antes haciendo los cálculos “a mano”.
8.2.17 Aditividad y linealidad
Cuando se plantea un modelo lineal, se supone implícitamente las condiciones siguientes:
- Aditividad: para cada variable independiente \(X_i\), la variación de asociada \(\mu_{Y|x_1,\ldots,x_k}\) con un aumento en \(X_i\) (manteniendo las otras variables constantes) es la misma sean cuales sean los valores de las otras variables independientes.
Dicho de otra forma: \[ \begin{array}{rl} \mu_{Y|x_1,\ldots,x_i+\Delta x_i,\ldots,x_k}-& \mu_{Y|x_1,\ldots,x_i,\ldots,x_k}=\\ & \beta_0+\beta_1 x_1+\cdots+ \beta_i (x_i+\Delta x_i)+\cdots + \beta_k x_k -\\ & (\beta_0+\beta_1 x_1+\cdots+\beta_i x_i+\cdots + \beta_k x_k) = \beta_i\Delta x_i. \end{array} \]
Fijémonos que la variación en el parámetro \(\mu_{\ldots}\) es independiente de los valores de \(x_1,\ldots,x_{i-1},x_{i+1},\ldots,x_k\).
- Linealidad: para cada variable independiente \(X_i\), la variación de asociada \(\mu_{Y|x_1,\ldots,x_k}\) con un aumento en \(X_i\) (manteniendo las otras variables constantes) es la misma sea cual sea el valor de \(X_i\).
Dicho de otra forma, si miramos la expresión anterior, la variación en el parámetro \(\mu_{\ldots}\) es independiente de los valores de \(x_i\).
8.2.18 Aditividad: test de Tukey
Podemos comprobar la aditividad con el test de Tukey, usando los llamados gráficos de residuos parciales para la linealidad.
La idea principal es verificar que no haya interacción entre las variables independientes y así, cada una tendrá un efecto aditivo en el modelo.
Si existe la interacción, algunos términos cuadráticos tendrán peso en el modelo. Esta es la base del Test de Tukey:
- Se obtienen los valores ajustados \(\{\hat{y}_i\}\) por la regresión lineal inicial.
- Se lleva a cabo una segunda regresión lineal incluyendo como nueva variable independiente los \(\hat{y}_i^2\). Sea \(\beta\) el coeficiente de esta nueva variable.
- Se testea si la variable \(\hat{y}^2\) es significativa en la segunda regresión. Es decir, se realiza el contraste \[\left.\begin{array}{ll} H_0: \beta=0,\\ H_1: \beta\neq 0.\end{array}\right\}\] Si no podemos descartar la hipótesis nula, la variable de los \(\hat{y}_i^2\) no es significativa y el modelo es aditivo.
Ejemplo de los bebés
Testeemos la aditividad para los datos del ejemplo de los bebés:
- Valores ajustados:
## 1 2 3 4 5 6 7 8 9
## 57.54 52.93 61.09 67.43 54.15 62.48 55.68 67.37 70.04- Segunda regresión lineal incluyendo los valores \(\hat{y}_i^2\):
valores.ajustados2 = reg.mul.original$fitted.values^2
summary(lm(y.bebes~X[,2]+X[,3]+X[,4]+X[,5]+valores.ajustados2))[[4]]## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -35.0865 35.50437 -0.9882 0.3959
## X[, 2] 0.4581 0.41441 1.1055 0.3496
## X[, 3] 1.9176 1.15881 1.6548 0.1965
## X[, 4] 8.8188 4.47436 1.9710 0.1433
## X[, 5] -0.1208 0.16794 -0.7195 0.5238
## valores.ajustados2 -0.0168 0.01277 -1.3151 0.2800Vemos que la variable \(\hat{y}_i^2\) no es significativa del modelo con un p-valor de 0.28. Concluimos por tanto que no tenemos evidencias suficientes para rechazar la aditividad del modelo.
R.
Para realizar el Test de Tukey en R hay que usar la función residualPlots del paquete car:
donde r es el objeto de R donde hemos guardado la información de la regresión original y plot es un parámetro que si vale TRUE (valor por defecto) nos dibuja los gráficos de los residuos frente a las variables regresoras y frente a los valores estimados junto con una curva de color azul indicando su tendencia y si vale FALSE simplemente da el valor del estadístico de Tukey y su p-valor.
Si optamos por ver los gráficos, no debe mostrarse ningún tipo de estructura, todos ellos deben parecer “cielos estrellados”.
Ejemplo de los bebés
Apliquemos el Test de Tukey para los datos del ejemplo de los bebés:
## Test stat Pr(>|Test stat|)
## X[, 2] -1.73 0.18
## X[, 3] -2.24 0.11
## X[, 4] -0.14 0.90
## X[, 5] -1.04 0.37
## Tukey test -1.32 0.19Obtenemos un p-valor grande, llegando a la misma conclusión que los cálculos realizados “a mano”.
8.2.19 Linealidad: gráficos de residuos parciales
Los gráficos de residuos parciales son una herramienta útil para detectar la no linealidad en una regresión.
Se definen los residuos parciales \(e_{ij}\) para la variable independiente \(X_j\) como \[e_{ij}=e_i+b_jx_{ij},\] donde \(e_i\) es el residuo \(i\)-ésimo de la regresión lineal, \(b_j\) es el coeficiente de \(X_j\) en la regresión original y \(x_{ij}\) es la observación \(j\)-ésima del individuo \(i\)-ésimo.
Los residuos parciales se dibujan contra los valores de \(x_j\) y se hace su recta de regresión.
Si ésta no se ajusta a la curva dada por una regresión no paramétrica suave (las variables independientes no están predeterminadas y se construyen con los datos), el modelo no es lineal.
La función de R para representar estos gráficos es crPlots del paquete car.
Ejemplo de los bebés
Realicemos los gráficos de residuos parciales para los datos del ejemplo de los bebés para ver gráficamente la linealidad:
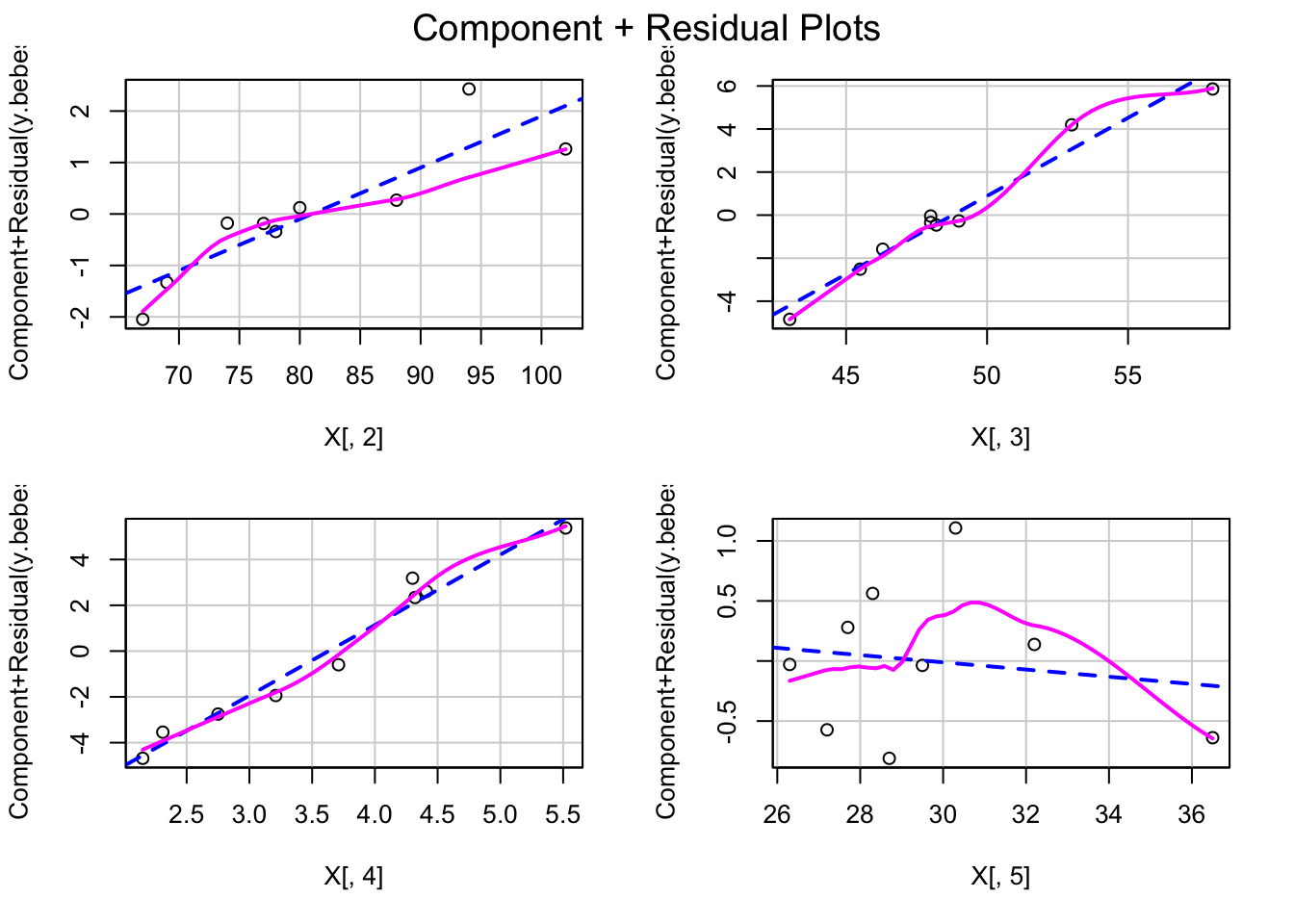
Observamos que la única variable que no se ajusta el modelo lineal es la variable \(x_4\): aumento en tanto por ciento de su peso actual respecto de su peso al nacer.
Todas las demás presentan un ajuste bastante aceptable al modelo lineal.
8.2.20 Observaciones anómalas
Las observaciones anómalas pueden provocar que se malinterpreten patrones en el conjunto de datos.
Además, puntos aislados pueden tener una gran influencia en el modelo de regresión dando resultados completamente diferentes.
Por ejemplo, pueden provocar que nuestro modelo no capture características importantes de los datos.
Por dichos motivos, es importante detectarlas.
Como se puede observar en el gráfico siguiente, la presencia de un valor anómalo distorsiona completamente el modelo.
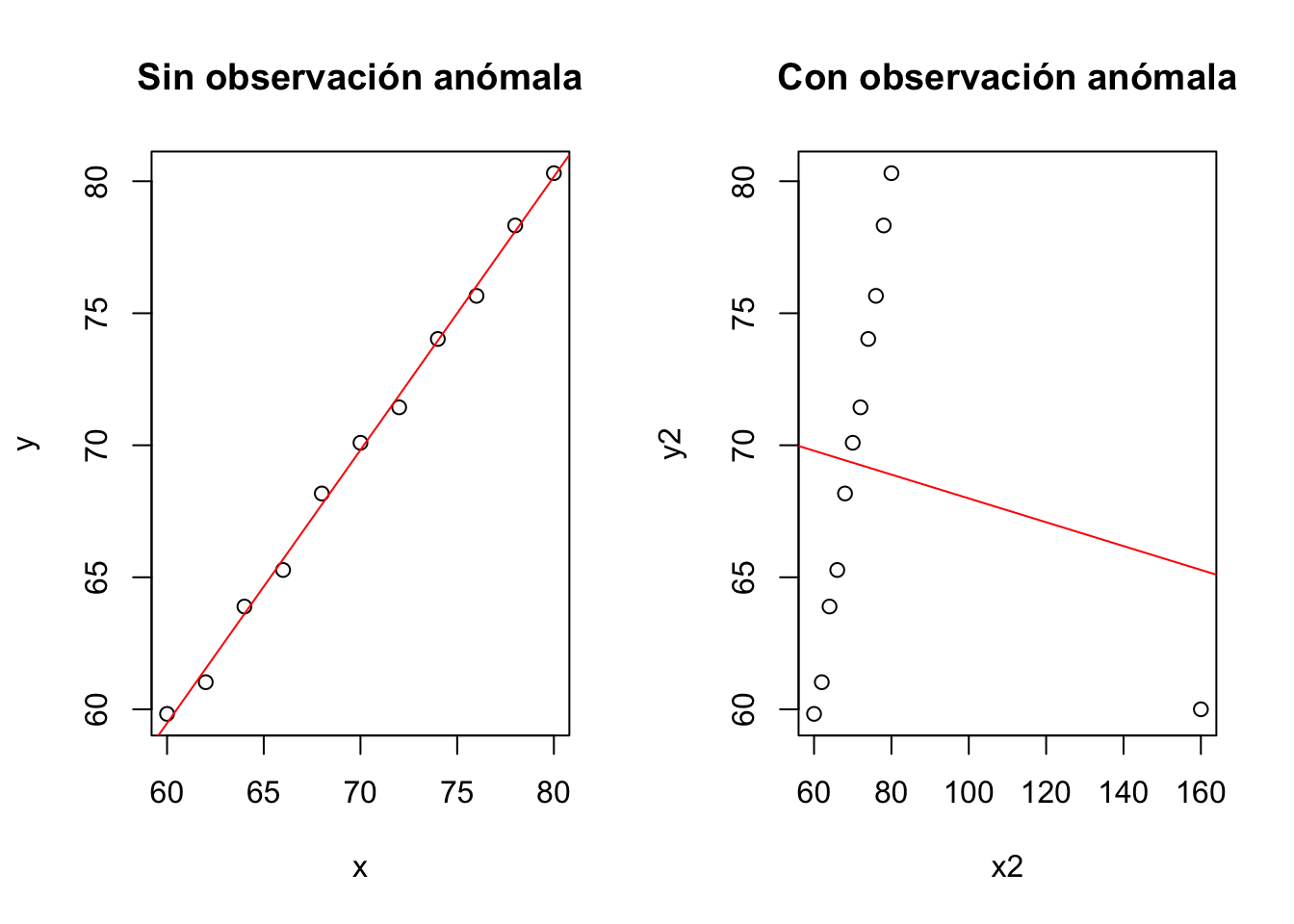
Existen tres tipos de observaciones anómalas:
- Outliers de regresión: son observaciones que tiene un valor anómalo de la variable dependiente \(Y\), condicionado a los valores de sus variables independientes \(X_i\). Tendrá un residuo muy alto pero puede no afectar demasiado a los coeficientes de la regresión.
- Leverages: son observaciones con un valor anómalo de las variables independientes \(X_i\). No tiene porqué afectar los coeficientes de la regresión.
- Observaciones influyentes: son aquellas que tienen un leverage alto, son outliers de regresión y afectan fuertemente a la regresión.
En el gráfico siguiente vemos un ejemplo de un outlier, un leverage y una observación anómala.
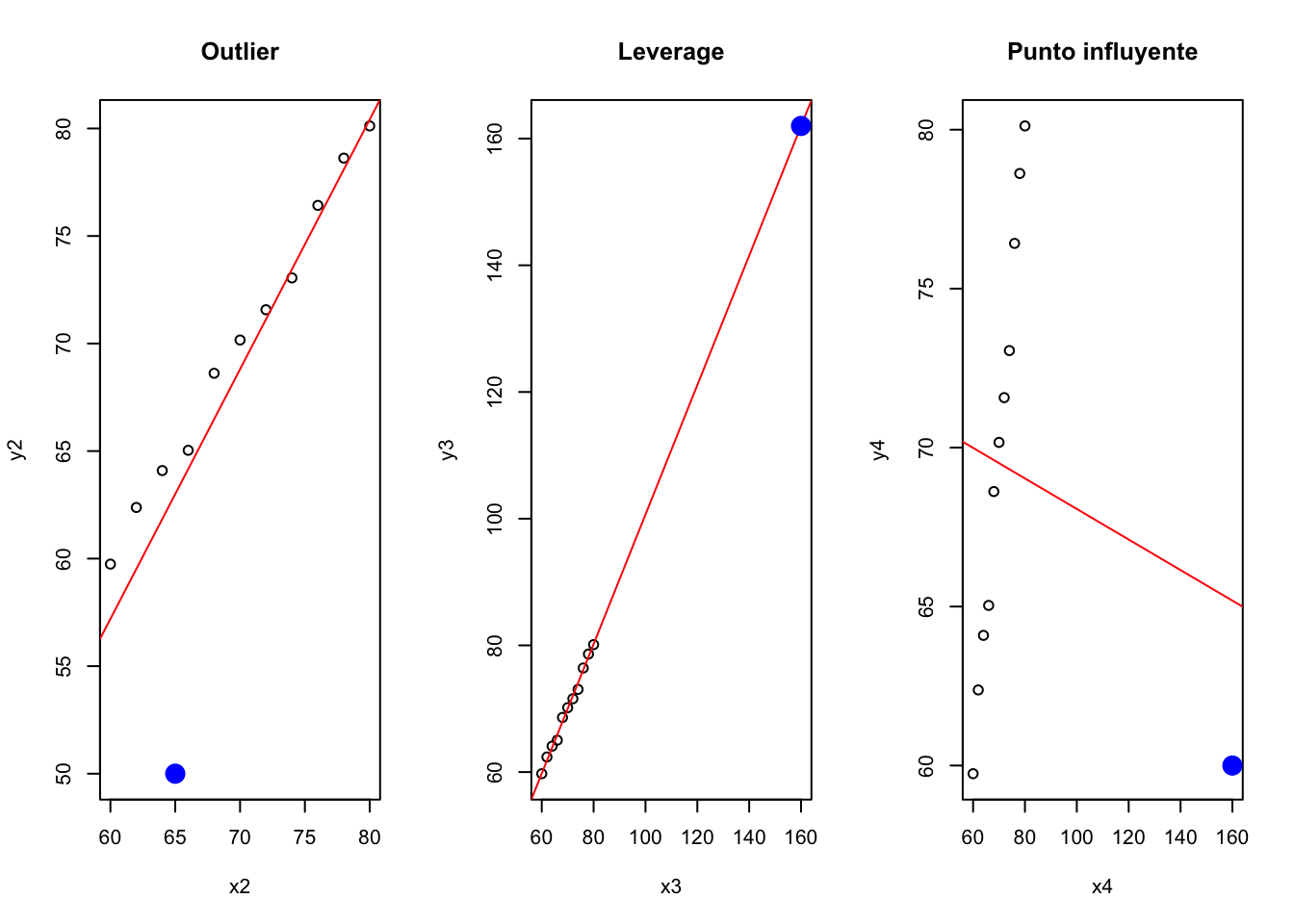
8.2.21 Leverages
Para hallar las observaciones que son leverages, en primer lugar, necesitamos definir la matriz Hat como: \[\mathbf{H}=\mathbf{X}(\mathbf{X}^\top \mathbf{X})^{-1}\mathbf{X}^\top.\] Esta matriz es simétrica (\(\mathbf{H}^\top=\mathbf{H}\)) e idempotente (\(\mathbf{H}^2=\mathbf{H}\)).
Además, es fácil comprobar que \[\hat{y}=\mathbf{X} b=\mathbf{X}(\mathbf{X}^\top \mathbf{X})^{-1}\mathbf{X}^\top y=\mathbf{H}y,\] usando la expresión dada anteriormente que nos hallaba los valores estimados \(\hat{y}\), y así tenemos que \(\hat{y}_{j}=h_{1j}y_1+h_{2j}y_2+\ldots+h_{nj}y_n=\sum_{i=1}^n{h_{ij}y_i},\) para \(j=1,\ldots,n\).
Si la componente \((i,j)\) de la matriz Hat, \(h_{ij}\) es grande, la observación \(i\)-ésima tiene un impacto sustancial en el valor predicho \(j\)-ésimo.
Se define el leverage de la observación \(i\)-ésima \(h_i\) como su valor hat \(h_i=\sum\limits_{j=1}^n{h_{ij}^2},\) y así, el valor hat \(h_i\) mide el leverage potencial de \(y_i\) en todos los valores predichos.
El valor hat medio es \(\overline{h}=\frac{1}{n}\sum\limits_{i=1}^n h_i=\frac{k+1}{n}\).
Los valores hat satisfacen \(\frac1{n}\leq h_i \leq 1\).
En la regresión lineal simple, los valores hat miden la distancia de \(x_i\) a la media de \(X\): \(h_i=\frac1{n}+\frac{(x_i-\overline{x})^2}{\sum\limits_{j=1}^n{(x_j-\overline{x} )^2}}.\)
En la regresión múltiple, \(h_i\) mide la distancia de una observación al vector medio de \(X\) de una forma parecida a la anterior.
Basándonos en las propiedades anteriores, la regla de decisión que nos dirá si una observación tiene leverage grande (y por lo tanto, tiene que ser considerada con cuidado) es cuando su valor hat cumpla: \(h_i>2\frac{k+1}{n}.\)
La función hatvalues de R calcula los valores hat dado un modelo de regresión basándose en la regla anterior:
donde r es el objeto de R donde hemos guardado la información de la regresión original.
Ejemplo de los bebés
Hallemos los valores leverages para la regresión realizada usando el ejemplo de los bebés:
## 1 2 3 4 5 6 7 8 9
## 0.4086 0.3615 0.3857 0.7313 0.5447 0.7008 0.6264 0.5139 0.7272## named integer(0)Vemos que en nuestro ejemplo no hay ninguna observación que puede considerarse con leverage alto.
8.2.22 Outliers
La estrategia para determinar qué observaciones son susceptibles de ser outliers se basan en los llamados residuos estunderizados.
Se basan en recalcular el modelo después de eliminar la observación \(i\)-ésima y hallar el correspondiente \((MSE)_i\).
Los residuos estunderizados se definen como: \[E_i^\star=\frac{e_i}{\sqrt{(MSE)_i(1-h_i)}},\] y, si el modelo es correcto, la variable anterior sigue una distribución \(t\) de Student con \(n-k-2\) grados de libertad.
Para detectar los outliers se siguen los pasos siguientes:
Para cada observación \(i\)-ésima, se calcula el residuo estunderizado \(E_i^\star\).
A continuación, se realiza una corrección de Bonferroni al p-valor multiplicándolo por \(n\) y así, el p-valor ajustado es \(2nP(t_{n-k-2}\geq E_i^\star).\)
Se van considerando por orden decreciente los p-valores de las observaciones hasta que se encuentra una observación que ya no sea un outlier.
La función de R que realiza este test de detección de outliers es la función outlierTest del paquete car.
donde r es el objeto de R donde hemos guardado la información de la regresión original.
Ejemplo de los bebés
Veamos si tenemos outliers para la regresión realizada usando el ejemplo de los bebés:
## No Studentized residuals with Bonferroni p < 0.05
## Largest |rstudent|:
## rstudent unadjusted p-value Bonferroni p
## 8 4.737 0.01784 0.1606Observamos que el bebé número 8 es el que tiene un residuo estunderizado más alto pero el p-valor ajustado de Bonferroni nos permite rechazar que sea un outlier.
8.2.23 Observaciones influyentes: Distancia de Cook
Como ya hemos indicado, una observación influyente es aquella que combina discrepancia con leverage.
Una forma de determinarlas es examinar cómo cambian los coeficientes de la regresión si se elimina una observación en concreto.
La medida para evaluar este cambio es la llamada distancia de Cook: \[D_i=\frac{e^2_{S_i}}{k+1}\cdot\frac{h_i}{1-h_i},\] dónde \(h_i\) es el leverage y es \(e_{S_i}\) el llamado residuo estandarizado, dado por \[e_{S_i}=\frac{e_i}{\sqrt{MSE(1-h_i)}}.\]
El primer factor en la expresión de la distancia de Cook (\(\frac{e^2_{S_i}}{k+1}\)) mide el grado de ser outlier mientras que el segundo (\(\frac{h_i}{1-h_i}\)) mide el grado de leverage.
Una regla para determinar qué observaciones son influyentes es \(D_i > \frac4{n-k-1}.\)
En R la distancia de Cook se calcula con la función cooks.distance del paquete car:
donde r es el objeto de R donde hemos guardado la información de la regresión original.
Ejemplo de los bebés
Calculemos las observaciones influyentes usando la distancia de Cook para la regresión realizada usando el ejemplo de los bebés:
## 1 2 3 4 5 6 7 8
## 0.0005175 0.0039792 0.0127434 0.5110093 0.2958675 0.1035268 0.3303470 0.7459674
## 9
## 1.8532517## 9
## 9Observamos que el bebé número 9 es una observación influyente según la distancia de Cook.
8.2.24 Tratamiento de las observaciones anómalas
El tratamiento de las observaciones anómalas es bastante complejo.
Nos debemos preguntar si se deben a errores en la entrada o recogida de los datos y si éste es el caso, se debían de eliminar.
Pero también pueden explicar que no se ha considerado alguna variable independiente que afecta al conjunto de observaciones anómalas.
Las más peligrosas son las influyentes.
En el supuesto de que se determine que se pueden eliminar, se tienen que eliminar de una a una, actualizando el modelo cada vez.
8.2.25 Algunas consideraciones finales: selección del modelo
El modelo de regresión lineal no es el único que podemos usar. Existen otros modelos como los polinómicos o los logarítmicos que podrían ajustar mejor nuestra tabla de datos.
El modelo puede ser más eficaz si añadimos otras variables, o puede ser igual de eficaz si eliminamos variables redundantes.
Puede haber dependencias lineales entre las variables que las haga redundantes. Podemos detectar dichas dependencias con la matriz de covarianzas entre las variables regresoras o independientes.
En R existe la función step que, a partir del método AIC nos da el mejor modelo desde en el sentido de buscar un equilibrio entre la simplicidad y la adecuación:
donde r es el objeto de R donde hemos guardado la información de la regresión original.
Ejemplo de los bebés
Si aplicamos la función step a la tabla de datos de los bebés, veamos cuál es el mejor modelo:
Con un AIC de -3.78, R nos dice que el mejor modelo es considerar las variables regresoras \(x_2\) (altura del bebé al nacer) y \(x_3\) (peso del bebé al nacer).
## Start: AIC=0.01
## y.bebes ~ X[, 2] + X[, 3] + X[, 4] + X[, 5]
##
## Df Sum of Sq RSS AIC
## - X[, 5] 1 0.02 2.99 -1.92
## - X[, 2] 1 0.06 3.03 -1.80
## - X[, 3] 1 0.63 3.60 -0.25
## <none> 2.97 0.01
## - X[, 4] 1 6.25 9.22 8.22
##
## Step: AIC=-1.92
## y.bebes ~ X[, 2] + X[, 3] + X[, 4]
##
## Df Sum of Sq RSS AIC
## - X[, 2] 1 0.05 3.04 -3.78
## <none> 2.99 -1.92
## - X[, 3] 1 0.79 3.78 -1.80
## - X[, 4] 1 6.23 9.22 6.22
##
## Step: AIC=-3.78
## y.bebes ~ X[, 3] + X[, 4]
##
## Df Sum of Sq RSS AIC
## <none> 3 -3.78
## - X[, 4] 1 103 106 26.19
## - X[, 3] 1 132 135 28.38##
## Call:
## lm(formula = y.bebes ~ X[, 3] + X[, 4])
##
## Coefficients:
## (Intercept) X[, 3] X[, 4]
## 2.183 0.958 3.3258.3 Guía rápida. Regresión lineal simple
lm(y~x): objeto dondeRguarda la información de la recta de regresión de la variableyen función de la variablex.summary(lm(y~x)): información detallada de la recta de regresión de la variableyen función de la variablex:summary(lm(y~x))$r.squared: nos da el coeficiente de determinación.summary(lm(y~x))$coefficients: nos da los coeficientes (\(b_0\) y \(b_1\)) de la recta de regresión, las desviaciones estándar del error de dichos estimadores, el valor \(t\) al hacer los contrastes de hipótesis para contrastar si son nulos y los p-valores correspondientes de dichos contrastes.
abline(lm(y~x)): dibuja la recta de regresión de la variableyen función de la variablexuna vez que se ha realizado un plot de la nube de puntos \((x_i,y_i)\), \(i=1,\ldots,n\).confint(lm(y~x),level=q): nos da el intervalo de confianza de los parámetros \(\beta_0\) y \(\beta_1\) al 100\(\cdot\)q% de confianza. Valor por defecto:q=0.95.predict.lm(lm(y~x),newdata,interval=...,level=q): da el intervalo de confianza para el parámetro \(\mu_{Y|x_0}\) o \(y_0\) al 100\(\cdot\)q% de confianza dependiendo de los valores del parámetrointerval: (newdatadebe ser undataframede una fila con el valor de la variable \(x_0\))interval="confidence": intervalo de confianza para \(\mu_{Y|x_0}\).interval="prediction": intervalo de confianza para \(y_0\).
8.4 Guía rápida. Regresión lineal múltiple
lm(y~x1+...xk): objeto dondeRguarda la información de la función de regresión de la variableyen función de las variables regresorasx1,\(\ldots\),xk.summary(lm(y~x1+...xk)): información detallada de la recta de regresión de la variableyen función de las variables regresorasx1,\(\ldots\),xk:summary(lm(y~x))$r.squared: nos da el coeficiente de determinación.summary(lm(y~x))$coefficients: nos da los coeficientes (\(b_0,b_1,\ldots,b_k\)) de la función de regresión, las desviaciones estándar del error de dichos estimadores, el valor \(t\) al hacer los contrastes de hipótesis para contrastar si son nulos y los p-valores correspondientes de dichos contrastes.summary(lm(y~x))$adj.r.squared: nos da el coeficiente de determinación ajustado.
AIC(lm(y~x1+...+xk)): nos da el valorAICdel modelo de regresión múltiplelm(y~x1+...xk).BIC(lm(y~x1+...+xk)): nos da el valorBICdel modelo de regresión múltiplelm(y~x1+...xk).confint(lm(y~x1+...+xk),level=q): nos da el intervalo de confianza de los parámetros \(b_0,b_1,\ldots,b_k\) al 100\(\cdot\)q% de confianza. Valor por defecto:q=0.95.predict.lm(lm(y~x1+...+xk),newdata,interval=...,level=q): da el intervalo de confianza para el parámetro \(\mu_{Y|x_{01},\ldots,x_{0k}}\) o \(y_0\) al 100\(\cdot\)q% de confianza dependiendo de los valores del parámetrointerval: (newdatadebe ser undataframede una fila con el valor de las variables \(x_{01},\ldots,x_{0k}\))interval="confidence": intervalo de confianza para \(\mu_{Y|x_{01},\ldots,x_{0k}}\).interval="prediction": intervalo de confianza para \(y_0\).
anova(lm(y~Xd)): calcula la tabla ANOVA de una regresión lineal múltiple deyen función de las columnas de la matrizXd, es decir,Xdes una matriz cuyas columnas son los valores de las variables independientes \(x_1,\ldots,x_k\).bptest(lm(y~x1+...+xk),~X+I(X^2))del paquetelmtest: realiza el test de White para verificar la homocedasticidad de los residuos en la regresión lineal múltiplelm(y~x1+...+xk)dondeXes la matriz que contiene los valores de la muestra de las variables independientes.bptest(lm(y~x1+...+xk)del paquetelmtest: realiza el test de Breuch-Pagan para verificar la homocedasticidad de los residuos en la regresión lineal múltiplelm(y~x1+...+xk).dwtest(lm(y~x1+...+xk,alternative=))del paquetelmtest: realiza el test de Durbin-Watson para comprobar la autocorrelación de los residuos donde si el parámetroalternativevale:greater: comprueba si los residuos tienen autocorrelación positiva.less: comprueba si los residuos tienen autocorrelación negativa.
residualPlot(lm(y~x1+...+xk),plot=...)del paquetecar: realiza el test de Tukey para verificar la aditividad del modelo lineal múltiplelm(y~x1+...+xk). Los valores del parámetroplotpueden ser:FALSE: da el valor del estadístico de contraste y el correspondiente p-valor.TRUE: dibuja los gráficos de los residuos frente a las variables regresoras \(x_1,\ldots, x_k\) y frente a los valores estimados \(\hat{y}_i\), \(i=1,\ldots,n\).
crPlots(lm(y~x1+...+xk))del paquetecar: realiza los gráficos de los residuos parciales para testear la linealidad del modelo de regresión múltiplelm(y~x1+...+xk).hatvalues(lm(y~x1+...+xk)): cálculos los valores hat para el modelo de regresión múltiplelm(y~x1+...+xk)con el objetivo de hallar las posibles observaciones leverages.outlierTest(lm(y~x1+...+xk)): realiza el test de detección de outliers para el modelo de regresión múltiplelm(y~x1+...+xk).cooks.distance(lm(y~x1+...+xk)): calcula las distancias de Cook de las observaciones para el modelo de regresión múltiplelm(y~x1+...+xk)con el fin de hallar las observaciones influyentes.step(lm(y~x1+...+xk)): da el mejor modelo de regresión múltiple en el sentido de buscar un equilibrio entre la simplicidad y la adecuación a partir del modelo completolm(y~x1+...+xk)usando el valorAIC.