Tema 7 Análisis de la Varianza
El problema que intentamos resolver es un contraste de igualdad de medias cuando tenemos más de dos poblaciones.
Concretamente, supongamos que \(k>2\) poblaciones.
Algunas veces, segregaremos la población por \(k\) subpoblaciones definidas por los niveles de un factor.
Por ejemplo, si queremos estudiar el peso de una población de una determinada edad, pongamos entre 30 y 40 años, podemos segregar por tipo de sangre, A, B, O y AB (\(k=4\)) y preguntarnos si el peso medio de cada subpoblación segredada por el tipo de sangre es el mismo.
Más concretamente, sean \(\mu_1,\ldots,\mu_k\) las medias de esta magnitud (peso en el ejemplo anterior) en cada una de las subpoblaciones o poblaciones. Nos planteamos el contraste siguiente: \[ \left\{ \begin{array}{l} H_0 : \mu_1 =\mu_2 =\cdots =\mu_k, \\ H_1 : \exists i,j \mid \mu_i \not=\mu_j. \end{array} \right. \]
Como es habitual, la decisión que tomaremos será en función de una muestra aleatoria de cada población.
La técnica que resuelve el contraste anterior se llama ANOVA de ANalysis Of VAriance en inglés, Análisis de la varianza, es castellano.
Esta técnica se puede aplicar bajo diferentes diseños de experimentos:
según cuántos factores usamos para separar la población en subpoblaciones,
según cómo escogemos los niveles de los factores,
según cómo escogemos las muestras.
Veremos los diseños más básicos. En un problema concreto, se tiene que decidir primero el tipo de experimento que se tiene que realizar.
Recordemos que en el caso de \(k=2\) poblaciones, para realizar la comparación de sus medias, calculábamos las medias de dos muestras y las comparábamos usando el estadístico de contraste correspondiente.
Si quisiéramos hacer lo mismo para \(k\geq 3\) poblaciones, tendríamos que comparar en total \(\dbinom{k}2\) pares de medias.
Para ver si hay diferencias, tenemos que realizar todas las comparaciones ya que podría pasar lo siguiente: \[ \mu_1\approx \mu_2, \mu_2\approx \mu_3, \mu_3\approx\mu_4\mbox{ pero } \mu_1\not\approx \mu_4 \]
Queremos un test que nos diga en un solo paso si todas son iguales, o si hay alguna diferente. Si hubiera diferencias, en una segunda fase, buscaríamos en qué pares de poblaciones hay medias diferentes.
El test ANOVA realiza la comparación de las medias de 3 o más poblaciones basándose en la variabilidad de los datos por grupos:
variabilidad de los datos (respecto de la media global),
variabilidad dentro de cada población (respecto de la media dentro de la población),
variabilidad de las medias por poblaciones (respecto de la media global).
La idea del test ANOVA es la siguiente: si la variabilidad total de los datos es explicada por la variabilidad de las medias de las poblaciones y la poca “variabilidad” dentro de cada población, es indicio que las medias son diferentes.
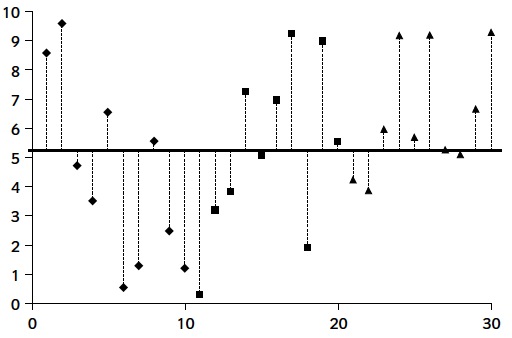
Las dos figuras siguientes muestra un ejemplo de tres muestras de tres poblaciones donde hay mucha variabilidad entre las medias de las muestras.
En cambio, hay poca variabilidad entre los valores dentro de cada muestra.
La media de la muestra de “rombos” está entorno al valor 9, la media de la muestra de “cuadrados”, entorno al valor 5 y la media de la muestra de “triángulos”, entorno al valor 1. Hay mucha diferencia entre las medias de las tres muestras.


En cambio en las dos figuras siguientes se muestra un ejemplo de tres muestras de tres poblaciones donde los valores tienen mucha variabilidad que no es explicada por la diferencia entre las medias de las muestras.
La media de la muestra de “rombos” está ahora entorno al valor 5.5, la media de la muestra de “cuadrados”, entorno al valor 5 y la media de la muestra de “triángulos”, entorno al valor 6. Vemos que en este caso, hay poca diferencia entre las medias de las tres muestras.

7.1 ANOVA de un factor
7.1.1 Clasificación simple, efectos fijos, diseño completamente aleatorio
Supongamos que estamos en los casos siguientes:
usamos un solo factor para clasificar la población en subpoblaciones (clasificación simple),
el investigador decide qué niveles (o tratamientos) del factor usará (efectos fijos),
se toma una m.a.s. de cada subpoblación, de manera independiente unas de las otras (completamente aleatorio)
Ejemplo: Pseudomonas fragi
Se realizó un estudio para investigar el efecto del CO\({}_2\) sobre la tasa de crecimiento de Pseudomonas fragi (un corruptor de alimentos). Se cree que el crecimiento se ve afectado por la cantidad de CO\({}_2\) en el aire.
Para contrastarlo, en un experimento se administró CO\({}_2\) a 5 presiones atmosféricas diferentes a 10 cultivos diferentes por cada nivel, y se anotó el cambio (en %) de la masa celular al cabo de una hora:
Presión de \(CO{}_2\) (en atmósferas)
| 0.0 | 0.083 | 0.29 | 0.50 | 0.86 |
|---|---|---|---|---|
| 62.6 | 50.9 | 45.5 | 29.5 | 24.9 |
| 59.6 | 44.3 | 41.1 | 22.8 | 17.2 |
| 64.5 | 47.5 | 29.8 | 19.2 | 7.8 |
| 59.3 | 49.5 | 38.3 | 20.6 | 10.5 |
| 58.6 | 48.5 | 40.2 | 29.2 | 17.8 |
| 64.6 | 50.4 | 38.5 | 24.1 | 22.1 |
| 50.9 | 35.2 | 30.2 | 22.6 | 22.6 |
| 56.2 | 49.9 | 27.0 | 32.7 | 16.8 |
| 52.3 | 42.6 | 40.0 | 24.4 | 15.9 |
| 62.8 | 41.6 | 33.9 | 29.6 | 8.8 |
7.1.2 Almacenamiento de datos en ANOVA
Los datos se suelen dar en forma de tabla donde las columnas suelen ser los niveles del factor.
Por tanto, en la columna \(i\)-ésima habrá la muestra correspondiente al factor \(i\)-ésimo.
Más concretamente, supongamos que los datos vienen con la estructura siguiente:
Niveles del factor
| \(F_1\) | \(F_2\) | \(\cdots\) | \(F_k\) |
|---|---|---|---|
| \(X_{11}\) | \(X_{21}\) | \(\cdots\) | \(X_{k1}\) |
| \(X_{12}\) | \(X_{22}\) | \(\cdots\) | \(X_{k2}\) |
| \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) |
| \(X_{1n_1}\) | \(X_{2n_2}\) | \(\cdots\) | \(X_{kn_k}\) |
donde
\(n_i\) es el tamaño de la muestra del nivel \(i\),
\(X_{ij}\) es el valor de la característica bajo estudio correspondiente al individuo \(j\) del nivel \(i\).
En el ejemplo anterior,
- \(k=5\),
- \(F_1=0.0\), \(F_2=0.083\), \(F_3=0.29\), \(F_4=0.50\) y \(F_5=0.86\),
- \(n_1=n_2=n_3=n_4=n_5=10\).
Para poder aplicar la técnica ANOVA, el paso previo es almacenar los datos en dos variables:
- variable característica, la llamaremos \(X\),
- variable factor, la llamaremos \(F\).
La variable \(X\) tendrá como valores los valores de la tabla anterior \(X_{ij}\) y la variable \(F\) valdrá \(i\) si el valor de la variable \(X\) corresponde al nivel \(i\)-ésimo del factor.
De esta manera transformaremos la tabla anterior en una tabla de \(N=n_1+\cdots + n_k\) filas y 2 columnas donde la primera columna serán los valores de la variable \(X\) y la segunda columna, los valores de la variable \(F\).
Ejemplo (continuación)
Realicemos la transformación anterior con los datos del ejemplo.
Llamaremos a la variable \(X\) Incremento.celular y a la variable \(F\), nivel.Presión.
Los valores de las variables anteriores serán:
Incremento.celular=c(
62.6,50.9,45.5,29.5,24.9,59.6,44.3,41.1,22.8,17.2,
64.5,47.5,29.8,19.2,7.8,59.3, 49.5,38.3,20.6,10.5,
58.6,48.5,40.2,29.2,17.8,64.6,50.4,38.5,24.1,22.1,
50.9,35.2,30.2,22.6,22.6,56.2,49.9,27.0,32.7,16.8,
52.3,42.6,40.0, 24.4,15.9,62.8,41.6,33.9,29.6,8.8)
nivel.Presión=rep(c("0.0","0.083","0.29","0.50","0.86"),times=10)Los primeros elementos de nuestra tabla transformada son:
## Incremento.celular nivel.Presión
## 1 62.6 0.0
## 2 50.9 0.083
## 3 45.5 0.29
## 4 29.5 0.50
## 5 24.9 0.86
## 6 59.6 0.0Fijémonos que hemos transformado la tabla original de 10 filas y 5 columnas en otra tabla de 50 filas y 2 columnas.
En general, si los datos vienen dados en una tabla donde las columnas representan los niveles de la variable factor, la tabla transformada tendrá \(N=n_1+\cdots +n_k\) filas y 2 columnas.
Ésta será la tabla sobre la que trabajaremos para realizar el contraste ANOVA.
Si realizamos un gráfico del porcentaje de aumento de masa celular según la variable presión del nivel de \(CO{}_2\), obtenemos lo siguiente:
presión = as.numeric(as.character(tabla.datos.ANOVA$nivel.Presión))
plot(presión,tabla.datos.ANOVA$Incremento.celular,type="p",
pch=18,cex=2,xlab="Nivel de CO2",ylab="% de masa celular")Observamos gráficamente que las medias del porcentaje del aumento de masa celular de las muestras correspondientes a los 5 niveles de presión parecen diferentes.

En el gráfico siguiente, dibujamos los 50 porcentajes de aumentos de masa celular correspondientes a los 50 microorganismos separándolos en 5 grupos correspondientes a los niveles de presión del nivel de \(CO{}_2\).
También aparecen las medias de cada grupo junto con la media global.
Este gráfico corrobora lo que hemos dicho anteriormente: parecen que hay diferencias entre las medias de los porcentajes de aumentos de masa celular entre los 5 grupos.
plot(1:50,tabla.datos.ANOVA$Incremento.celular,type="p",pch=17,
xlab="Número de microorganismo (por nivel)",ylab="% de incremento de masa celular",cex=0)
points(1:10,tabla.datos.ANOVA$Incremento.celular[seq(from=1,to=46,by=5)],pch=18,cex=1.5)
points(11:20,tabla.datos.ANOVA$Incremento.celular[seq(from=2,to=47,by=5)],pch=15,cex=1.5)
points(21:30,tabla.datos.ANOVA$Incremento.celular[seq(from=3,to=48,by=5)],pch=17,cex=1.5)
points(31:40,tabla.datos.ANOVA$Incremento.celular[seq(from=4,to=49,by=5)],pch=19,cex=1.5)
points(41:50,tabla.datos.ANOVA$Incremento.celular[seq(from=5,to=50,by=5)],pch=8,cex=1.5)
lines(c(0,50),c(mean(tabla.datos.ANOVA$Incremento.celular),
mean(mean(c(tabla.datos.ANOVA$Incremento.celular)))),lwd=3,col="red")
text(5,mean(mean(tabla.datos.ANOVA$Incremento.celular)-2),"Media global",col="red")
for (i in 1:5){lines(c(0+10*(i-1),10*i),
c(mean(tabla.datos.ANOVA$Incremento.celular[seq(from=i,to=46+i,by=5)]),
mean(tabla.datos.ANOVA$Incremento.celular[seq(from=i,to=46+i,by=5)])),lwd=3)}
text(20,mean(tabla.datos.ANOVA$Incremento.celular[seq(from=1,to=46,by=5)]),
"Media del Nivel 0.0")
text(30,mean(tabla.datos.ANOVA$Incremento.celular[seq(from=2,to=47,by=5)]),
"Media del Nivel 0.083")
text(40,mean(tabla.datos.ANOVA$Incremento.celular[seq(from=3,to=48,by=5)]-1),
"Media del Nivel 0.29")
text(20,mean(tabla.datos.ANOVA$Incremento.celular[seq(from=4,to=49,by=5)]),
"Media del Nivel 0.50")
text(30,mean(tabla.datos.ANOVA$Incremento.celular[seq(from=5,to=50,by=5)]),
"Media del Nivel 0.86")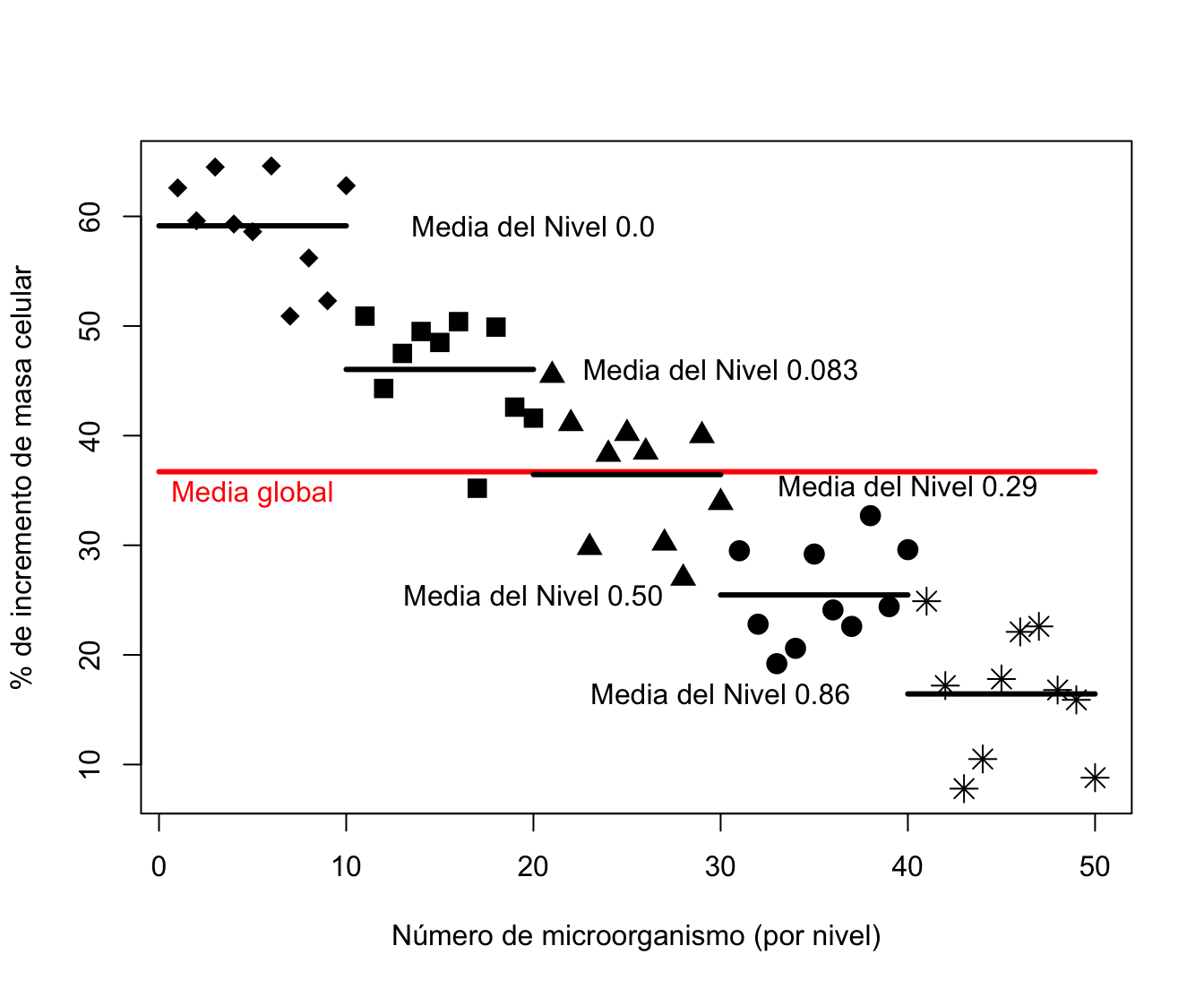
7.1.3 El contraste ANOVA
Recordemos que los datos vienen dados según la estructura siguiente:
Niveles del factor
| \(F_1\) | \(F_2\) | \(\cdots\) | \(F_k\) |
|---|---|---|---|
| \(X_{11}\) | \(X_{21}\) | \(\cdots\) | \(X_{k1}\) |
| \(X_{12}\) | \(X_{22}\) | \(\cdots\) | \(X_{k2}\) |
| \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) |
| \(X_{1n_1}\) | \(X_{2n_2}\) | \(\cdots\) | \(X_{kn_k}\) |
donde
\(n_i\) es el tamaño de la muestra del nivel \(i\),
\(X_{ij}\) es el valor de la característica bajo estudio correspondiente al individuo \(j\) del nivel \(i\).
7.1.4 Estadísticos
A partir de los datos de las muestras, definimos los estadísticos siguientes:
- Suma total de los datos del nivel \(i\)-ésimo: \(\displaystyle T_{i\bullet} =\sum_{j=1}^{n_i} X_{ij}.\)
- Media muestral para el nivel \(i\)-ésimo: \(\displaystyle\overline{X}_{i\bullet} = \frac{T_{i\bullet}}{n_i}.\)
- Suma total de los datos: \(\displaystyle T_{\bullet\bullet}=\sum_{i=1}^k\sum_{j=1}^{n_i} X_{ij} =\sum_{i=1}^k T_{i\bullet}.\)
- Media muestral de todos los datos: \(\displaystyle \overline{X}_{\bullet\bullet} =\frac{T_{\bullet\bullet}}{N},\) donde \(N=n_1+\cdots + n_k.\)
Calculemos los estadísticos anteriores para los datos de nuestro ejemplo.
- Para calcular la suma total de los datos del \(i\)-ésimo nivel, usaremos la función
aggregatedeR:
sumas.niveles = aggregate(Incremento.celular ~ nivel.Presión,
data = tabla.datos.ANOVA, FUN="sum")
sumas.niveles## nivel.Presión Incremento.celular
## 1 0.0 591.4
## 2 0.083 460.4
## 3 0.29 364.5
## 4 0.50 254.7
## 5 0.86 164.4- Para calcular la media total de los datos del \(i\)-ésimo nivel, podemos usar otra vez la función
aggregatedeR:
medias.niveles = aggregate(Incremento.celular ~ nivel.Presión,
data=tabla.datos.ANOVA, FUN="mean")
medias.niveles## nivel.Presión Incremento.celular
## 1 0.0 59.14
## 2 0.083 46.04
## 3 0.29 36.45
## 4 0.50 25.47
## 5 0.86 16.44- La suma total de los datos será:
## [1] 1835- La media muestral de los datos será:
## [1] 36.717.1.5 El modelo
Los parámetros que intervendrán en el contraste son:
\(\mu\): media poblacional del conjunto de la población (ignorando los niveles).
\(\mu_i\): media poblacional dentro del nivel \(i\)-ésimo, \(i=1,\ldots,k\).
Los estimadores de los parámetros son los siguientes:
De: \(\mu\), \(\overline{X}_{\bullet\bullet}\).
De cada \(\mu_i\), \(\overline{X}_{i\bullet}\).
Las suposiciones del modelo son:
Las \(k\) muestras son m.a.s. independientes extraídas de \(k\) poblaciones específicas con medias \(\mu_1,\ldots,\mu_k\).
Cada una de las \(k\) poblaciones sigue una ley normal.
Todas estas poblaciones tienen la misma varianza \(\sigma^2\) (homocedasticidad).
La expresión matemática del modelo a estudiar consiste en separar las diferencias de los datos respecto la media global en dos sumandos: las diferencias de los datos respecto las medias de cada nivel y las diferencias de las medias de cada nivel respecto la media global: \[ X_{ij} - \mu = (X_{ij} -\mu_i)+(\mu_i - \mu),\ i=1,\ldots,k,\ j=1,\ldots,n_i, \] dónde
\(X_{ij}\): valor del \(j\)-ésimo individuo dentro del nivel \(i\)-ésimo,
\(X_{ij}-\mu\): desviación del individuo respecto de la media global,
\(X_{ij}-\mu_i\): desviación del individuo respecto de la media de su grupo,
\(\mu_i -\mu\): desviación de la media del grupo \(i\)-ésimo respecto de la media global.
7.1.6 Identidad de la suma de cuadrados
El teorema siguiente es la clave del contraste ANOVA.
Separa la variabilidad de los datos respecto la media global denominada Suma Total de Cuadrados en dos variabilidades:
La variabilidad de las medias de cada grupo respecto la media global. A dicha variabilidad la llamaremos Suma de Cuadrados de los Tratamientos. Interesa que dicha variabilidad sea pequeña para que la hipótesis nula de igualdad de medias sea cierta.
La variabilidad de los datos respecto la media de cada grupo. A dicha variabilidad la llamaremos Suma de Cuadrados de los Residuos o Errores. Interesa que dicha variabilidad sea grande para tener grupos lo más heterogéneos posibles de cara a aumentar la potencia del contraste ANOVA.
\[\displaystyle \sum_{i=1}^k\sum_{j=1}^{n_i} (X_{ij}-\overline{X}_{\bullet\bullet})^2 = \sum_{i=1}^k n_i (\overline{X}_{i\bullet}-\overline{X}_{\bullet\bullet})^2 + \sum_{i=1}^k\sum_{j=1}^{n_i} (X_{ij}-\overline{X}_{i\bullet})^2\]
\(SS_{Total}=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} (X_{ij}-\overline{X}_{\bullet\bullet})^2\) (Suma Total de Cuadrados)
\(SS_{Tr}=\sum\limits_{i=1}^k n_i (\overline{X}_{i\bullet}-\overline{X}_{\bullet\bullet})^2\) (Suma de Cuadrados de los Tratamientos)
\(SS_E=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} (X_{ij}-\overline{X}_{i\bullet})^2\) (Suma de Cuadrados de los Residuos o Errores)
De cara a calcular las variabilidades o las sumas anteriores podemos usar las siguientes fórmulas equivalentes:
\(\displaystyle SS_{Total}=\sum_{i=1}^k\sum_{j=1}^{n_i} X_{ij}^2 - \frac{T_{\bullet\bullet}^2}{N} = T^{(2)}_{\bullet\bullet} - \frac{T_{\bullet\bullet}^2}{N}\).
\(\displaystyle SS_{Tr}=\sum_{i=1}^k \frac{T_{i\bullet}^2}{n_i} -\frac{T_{\bullet\bullet}^2}{N}\).
\(SS_E=SS_{Total}-SS_{Tr}\).
Ejercicio
Demostrar las fórmulas anteriores.
Usualmente escribiremos, para abreviar, \[ T^{(2)}_{\bullet\bullet}=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} X_{ij}^2. \]
Calculemos las variabilidades o sumas anteriores para los datos de nuestro ejemplo de dos maneras distintas tal como queda indicado en las expresiones. Nos vamos a ayudar de las variables siguientes:
##
## 0.0 0.083 0.29 0.50 0.86
## 10 10 10 10 10## [1] 50- \(SS_{Total}=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} (X_{ij}-\overline{X}_{\bullet\bullet})^2=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} X_{ij}^2 - \dfrac{T_{\bullet\bullet}^2}{N}\):
## [1] 12522## [1] 12522- \(SS_{Tr}=\sum\limits_{i=1}^k n_i (\overline{X}_{i\bullet}-\overline{X}_{\bullet\bullet})^2=\sum\limits_{i=1}^k \dfrac{T_{i\bullet}^2}{n_i} -\dfrac{T_{\bullet\bullet}^2}{N}\):
## [1] 11274## [1] 11274- \(SS_E=\sum\limits_{i=1}^k\sum\limits_{j=1}^{n_i} (X_{ij}-\overline{X}_{i\bullet})^2=SS_{Total}-SS_{Tr}\):
## [1] 1248## [1] 12487.1.7 Estadísticos del contraste
Usaremos los estadísticos de contraste siguientes:
Cuadrado medio de los tratamientos: \[ MS_{Tr}=\frac{SS_{Tr}}{k-1}. \]
Cuadrado medio residual: \[ MS_E=\frac{SS_E}{N-k}. \]
Estos estadísticos son variables aleatorias, y se tiene que
\(E(MS_{Tr})=\displaystyle\sigma^2 + \sum_{i=1}^k \frac{n_i (\mu_i -\mu)^2}{k-1}\).
\(E(MS_E)=\sigma^2\).
En particular, se puede usar \(MS_E\) para estimar la varianza común \(\sigma^2\).
Si la hipótesis nula \(H_0:\mu_1=\cdots=\mu_k=\mu\) es cierta, tenemos la siguiente condición: \[ \sum_{i=1}^k \frac{n_i (\mu_i -\mu)^2}{k-1}=0, \] y si \(H_0\) no es cierta, esta cantidad es estrictamente positiva.
Por lo tanto
- si la hipótesis nula \(H_0\) es cierta, \(E(MS_E)=E(MS_{Tr})\) y consecuentemente tendríamos que esperar que estos dos estadísticos tuvieran valores parecidos, es decir \[ \frac{MS_{Tr}}{MS_E}\approx 1. \]
- si la hipótesis nula \(H_0\) es falsa, \(E(MS_E)<E(MS_{Tr})\) y consecuentemente tendríamos que esperar que \[ \frac{MS_{Tr}}{MS_E}> 1. \]
Basándonos en las consideraciones anteriores, consideraremos como estadístico de contraste el cociente \[ F=\frac{MS_{Tr}}{MS_E}, \] que, si la hipótesis nula \(H_0\) es cierta, se distribuye según una \(F_{k-1,N-k}\) (F de Fisher con \(k-1\) y grados \(N-k\) de libertad).
Su valor será cercano a 1. Por tanto, rechazaremos la hipótesis nula si \(F\) es “bastante más grande” que 1.
Sinteticemos cómo realizar el contraste ANOVA en 4 pasos:
Primer paso: calculamos las sumas de cuadrados: \(SS_{Total}, SS_{Tr}, SS_E.\)
Segundo paso: calculamos los cuadrados medios: \(MS_{Tr}=\frac{SS_{Tr}}{k-1},\ MS_E=\frac{SS_E}{N-k}.\)
Tercer paso: calculamos el estadístico de contraste \(F\): \(F=\frac{MS_{Tr}}{MS_E}.\)
Cuarto paso: calculamos el p-valor del contraste: \(P(F_{k-1,N-k}\geq F)\). Si dicho p-valor es más pequeño que el nivel de significación \(\alpha\), rechazamos \(H_0\) y concluimos que no todas las medias son iguales. En caso contrario, aceptamos \(H_0\).
Realicemos el contraste ANOVA para los datos de nuestro ejemplo.
- Sumas de cuadrados:
## [1] 12522## [1] 11274## [1] 1248- Cuadrados medios:
## [1] 2819## [1] 27.73- Estadístico de contraste:
## [1] 101.6- p-valor:
## [1] 0.0000000000000000000006233Comprobamos que el p-valor es muy pequeño. Concluimos, por tanto, que tenemos evidencias suficientes para concluir que las medias del aumento de masa celular no son iguales para los diferentes niveles de presión de \(CO{}_2\).
Y, consecuentemente, el nivel de presión de CO\({}_2\) puede influir en el crecimiento del microorganismo Pseudomonas fragi.
7.1.8 Tabla ANOVA
El contraste ANOVA se resume en la tabla siguiente:
| Origen variación | Grados de libertad | Sumas de cuadrados | Cuadrados medios | Estadístico de contraste | p-valor |
|---|---|---|---|---|---|
| Nivel | \(k-1\) | \(SS_{Tr}\) | \(MS_{Tr}=\frac{SS_{Tr}}{k-1}\) | \(F=\frac{MS_{Tr}}{MS_E}\) | p-valor |
| Residuo | \(N-k\) | \(SS_E\) | \(MS_E=\frac{SS_E}{N-k}\) |
La tabla anterior para los datos de nuestro ejemplo es la siguiente:
| Origen variación | Grados de libertad | Sumas de cuadrados | Cuadrados medios | Estadístico de contraste | p-valor |
|---|---|---|---|---|---|
| Nivel | 4 | 11274.32 | 2818.58 | 101.63 | 0 |
| Residuo | 45 | 1248.04 | 27.73 |
7.1.9 Contraste ANOVA con R
Para realizar un contraste ANOVA en R hay que usar la función aov.
Dicha función se aplica a la tabla de datos modificada que hemos explicado:
donde recordemos que en la variable X se almacenan los valores \(X_{ij}\) y en la variable factor F, los niveles del factor.
Lo primero que podríamos hacer es visualizar los datos del ejemplo con un boxplot:
boxplot(Incremento.celular ~ nivel.Presión, data = tabla.datos.ANOVA,
xlab="Presión",ylab="Incremento celular")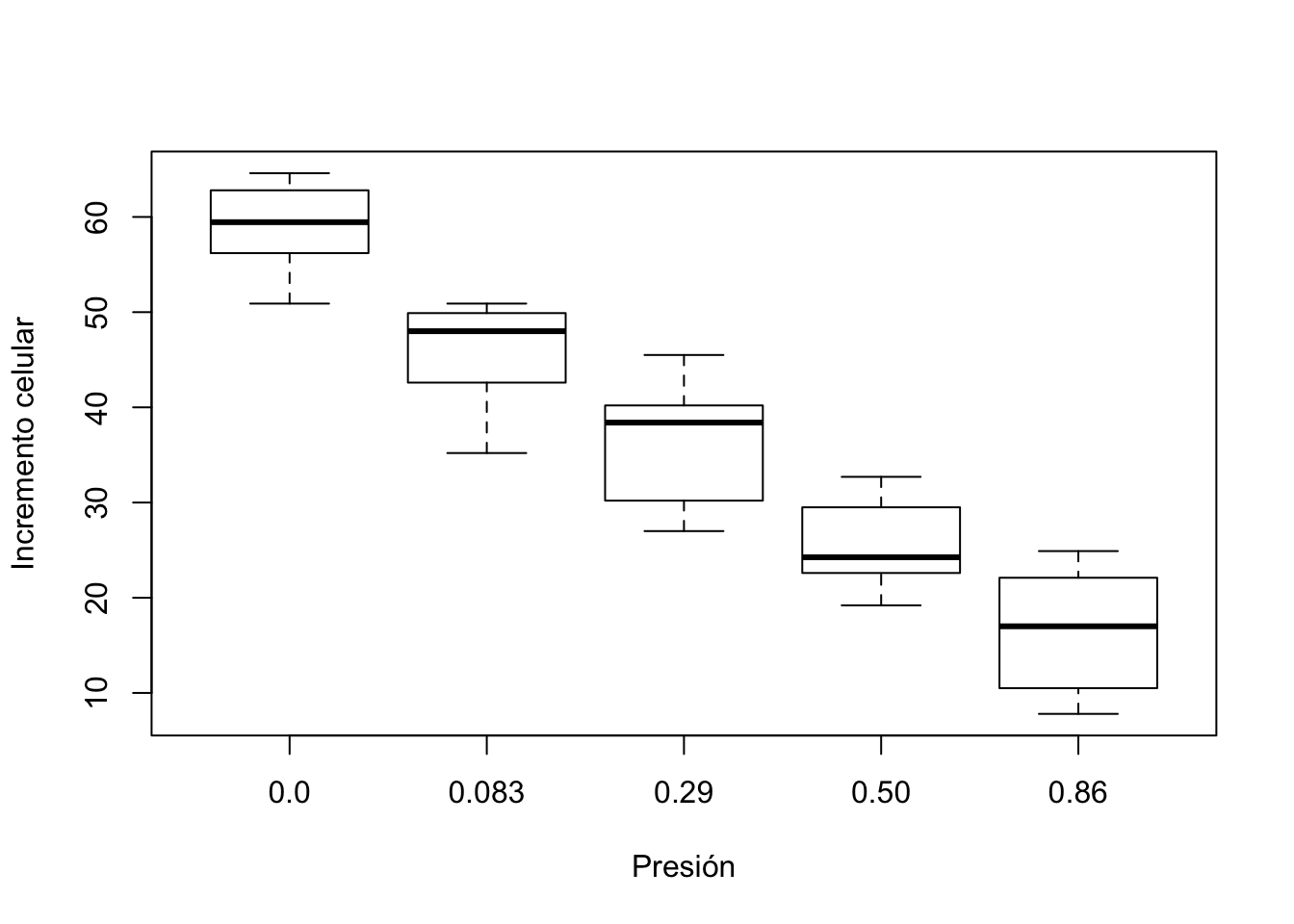
Vemos que gráficamente se observan diferencias para los distintos niveles de presión.
El contraste ANOVA en R se realizaría de la forma siguiente:
## Df Sum Sq Mean Sq F value Pr(>F)
## F 4 11274 2819 102 <0.0000000000000002 ***
## Residuals 45 1248 28
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Observamos que tenemos la misma tabla ANOVA obtenida haciendo los cálculos “a mano”.
7.2 Comparaciones por parejas
Si hemos rechazado la hipótesis nula \(H_0:\mu_1=\cdots =\mu_k\), el siguiente paso es averiguar cuáles son los niveles diferentes.
Es decir, hallar aquellas parejas \((\mu_i,\mu_j)\) para las que podamos decir que \(\mu_i\neq \mu_j\).
Aunque hay diferentes formas de hacerlo vamos a ver las más usuales.
7.2.1 Test T de Bonferroni
Hay que tener en cuenta que hay que realizar en total \(\displaystyle\binom{k}2\) contrastes del tipo: \[ \left. \begin{array}{ll} H_0 &: \mu_i=\mu_j, \\ H_1 &: \mu_i\not=\mu_j. \end{array} \right\} \] El estadístico de cada contraste es el siguiente: \[ T=\frac{\overline{X}_{i\bullet} - \overline{X}_{j\bullet}}{\sqrt{MS_E\cdot (\frac1{n_i} +\frac1{n_j})}}, \] que, si la hipótesis nula \(H_0\) es cierta, sigue una distribución \(t\) de Student con \(N-k\) grados de libertad, \(t_{N-k}\).
El \(p\)-valor de cada contraste es \(2P(t_{N-k}\geq |t_{i,j}|)\), donde \(t_{i,j}\) es el valor que toma el estadístico.
En el ejemplo del aumento de la masa corporal del microorganismo, si realizamos \(c=\binom52=10\) contrastes con nivel de significación \(\alpha =0.05\), ¡la probabilidad de Error de Tipo I en al menos uno de ellos es \(1-(1-0.05)^{10} \approx 0.4\)!
Por tanto, tendremos que reducir el nivel de significación de cada contraste para que la probabilidad final de Error de Tipo I sea \(\alpha\).
Usaremos la aproximación \(1-(1-x)^c \approx cx\) y entonces, si queremos efectuar \(c\) contrastes con nivel de significación (global) \(\alpha\), los haremos con nivel de significación \(\alpha/c\).
En el ejemplo del aumento de la masa corporal del microorganismo, si realizamos los \(10\) contrastes, para obtener un nivel de significación global \(\alpha =0.05\), realizamos cada contraste con nivel de significación \(\frac{0.05}{10}=0.005\).
Realicemos el test de Bonferroni para los datos de nuestro ejemplo.
En primer lugar creamos una matriz cuyas filas son las parejas de niveles las medias de los cuales contrastaremos:
## [,1] [,2]
## [1,] 1 2
## [2,] 1 3
## [3,] 1 4
## [4,] 1 5
## [5,] 2 3
## [6,] 2 4
## [7,] 2 5
## [8,] 3 4
## [9,] 3 5
## [10,] 4 5A continuación calculamos los valores de todos los estadísticos de contraste:
\[ T=\frac{\overline{X}_{i\bullet} - \overline{X}_{j\bullet}}{\sqrt{MS_E\cdot (\frac1{n_i} +\frac1{n_j})}}, \]
(est.contraste.pares = (medias.niveles[pares[,1],2]-medias.niveles[pares[,2],2])/
(sqrt(MSE*(1/10+1/10))))## [1] 5.562 9.634 14.296 18.130 4.072 8.734 12.568 4.662 8.496 3.834Los añadimos como columna a la matriz de parejas de niveles:
## est.contraste.pares
## [1,] 1 2 5.562
## [2,] 1 3 9.634
## [3,] 1 4 14.296
## [4,] 1 5 18.130
## [5,] 2 3 4.072
## [6,] 2 4 8.734
## [7,] 2 5 12.568
## [8,] 3 4 4.662
## [9,] 3 5 8.496
## [10,] 4 5 3.834Calculamos los p-valores:
calculo.p.valor=function(x){2*(1-pt(abs(x),N-k))}
(p.valores=sapply(est.contraste.pares,calculo.p.valor))## [1] 0.000001387522490681 0.000000000001649791 0.000000000000000000
## [4] 0.000000000000000000 0.000186374413800872 0.000000000030210501
## [7] 0.000000000000000222 0.000028080321327284 0.000000000066096462
## [10] 0.000389221804025119Lo añadimos como columna a la matriz de parejas de niveles y estadísticos:
## est.contraste.pares p.valores
## [1,] 1 2 5.562 0.000001387522490681
## [2,] 1 3 9.634 0.000000000001649791
## [3,] 1 4 14.296 0.000000000000000000
## [4,] 1 5 18.130 0.000000000000000000
## [5,] 2 3 4.072 0.000186374413800872
## [6,] 2 4 8.734 0.000000000030210501
## [7,] 2 5 12.568 0.000000000000000222
## [8,] 3 4 4.662 0.000028080321327284
## [9,] 3 5 8.496 0.000000000066096462
## [10,] 4 5 3.834 0.000389221804025119A continuación nos preguntamos qué p-valores son menores que 0.05/10 para saber entre qué pares podemos aceptar que hay diferencias:
## est.contraste.pares p.valores
## [1,] 1 2 5.562 0.000001387522490681
## [2,] 1 3 9.634 0.000000000001649791
## [3,] 1 4 14.296 0.000000000000000000
## [4,] 1 5 18.130 0.000000000000000000
## [5,] 2 3 4.072 0.000186374413800872
## [6,] 2 4 8.734 0.000000000030210501
## [7,] 2 5 12.568 0.000000000000000222
## [8,] 3 4 4.662 0.000028080321327284
## [9,] 3 5 8.496 0.000000000066096462
## [10,] 4 5 3.834 0.000389221804025119Vemos que en todos los pares se verifica que el p-valor es menor que 0.005.
Concluimos que hay evidencias suficientes para rechazar la igualdad de medias de aumento de masa celular del microorganismo entre dos niveles cualquiera de presión de \(CO{}_2\). Por tanto, \(\mu_i\neq \mu_j\) para todo \(i\neq j\).
Para realizar la comparación por parejas con R, usamos la función pairwise.t.test. El parámetro p.adjust.method puede tomar el valor “none” (que no hace ajuste alguno):
##
## Pairwise comparisons using t tests with pooled SD
##
## data: X and F
##
## 0.0 0.083 0.29
## 0.083 0.0000013875224908 - -
## 0.29 0.0000000000016497 0.0001863744138008 -
## 0.50 < 0.0000000000000002 0.0000000000302105 0.0000280803213273
## 0.86 < 0.0000000000000002 0.0000000000000003 0.0000000000660966
## 0.50
## 0.083 -
## 0.29 -
## 0.50 -
## 0.86 0.0003892218040251
##
## P value adjustment method: noneO bien el parámetro p.adjust.method puede tomar el valor “bonferroni” (que multiplicará el p-valor del contraste por el número de comparaciones llevadas a cabo, \(\binom{k}{2}\)):
##
## Pairwise comparisons using t tests with pooled SD
##
## data: X and F
##
## 0.0 0.083 0.29 0.50
## 0.083 0.000013875224908 - - -
## 0.29 0.000000000016497 0.002 - -
## 0.50 < 0.0000000000000002 0.000000000302105 0.000280803213273 -
## 0.86 < 0.0000000000000002 0.000000000000003 0.000000000660966 0.004
##
## P value adjustment method: bonferroniR nos muestra una tabla donde las filas y las columnas son los niveles del factor y cuyos valores son los p-valores de los contrastes por parejas.
Observamos que, en el primer caso todos los p-valores son menores que \(\alpha/c=0.05/10\) (sin hacer ningún ajuste en el método) o bien que en el segundo caso, todos los p-valores son menores que \(\alpha=0.05\), llegando en ambos casos a la misma conclusión que antes.
7.2.2 Test T de Holm (más potente)
El test de Holm es otro método que nos permite realizar las comparaciones entre las parejas de los distintos niveles del factor.
Dicho método es más usado ya que tiene más potencia que el método de Bonferroni.
Consta de los pasos siguientes:
Sean \(C_1,\ldots ,C_{c}\) los contrastes y los \(P_1,\ldots ,P_{c}\) p-valores correspondientes
Ordenamos estos \(p\)-valores en orden creciente \(P_{(1)}\leq \cdots\leq P_{(c)}\) y reenumeramos consistentemente los contrastes \(C_{(1)},\ldots, C_{(c)}\).
Para cada \(j=1,\ldots,c\), calculamos el p-valor ajustado \(\widetilde{P} _{(j)}=(c+1-j)P_{(j)}\).
Entonces rechazamos la hipótesis nula del contraste \(C_{(j)}\) si \(\widetilde{P}_{(j)}<\alpha\).
Vamos a aplicar el método de Holm al ejemplo que hemos estado desarrollando.
En primer lugar, ordenamos las filas de la tabla de datos pares ordenando los p-valores de menor a mayor:
## est.contraste.pares p.valores
## [1,] 1 4 14.296 0.000000000000000000
## [2,] 1 5 18.130 0.000000000000000000
## [3,] 2 5 12.568 0.000000000000000222
## [4,] 1 3 9.634 0.000000000001649791
## [5,] 2 4 8.734 0.000000000030210501
## [6,] 3 5 8.496 0.000000000066096462
## [7,] 1 2 5.562 0.000001387522490681
## [8,] 3 4 4.662 0.000028080321327284
## [9,] 2 3 4.072 0.000186374413800872
## [10,] 4 5 3.834 0.000389221804025119Calculamos los p-valores ajustados y los añadimos como columna a pares.ord:
p.valores.ajust=pares.ord[,4]*(10+1-1:10)
pares.ord=cbind(pares.ord,p.valores.ajust)
round(pares.ord,12)## est.contraste.pares p.valores p.valores.ajust
## [1,] 1 4 14.296 0.000000000000 0.000000000000
## [2,] 1 5 18.130 0.000000000000 0.000000000000
## [3,] 2 5 12.568 0.000000000000 0.000000000000
## [4,] 1 3 9.634 0.000000000002 0.000000000012
## [5,] 2 4 8.734 0.000000000030 0.000000000181
## [6,] 3 5 8.496 0.000000000066 0.000000000330
## [7,] 1 2 5.562 0.000001387522 0.000005550090
## [8,] 3 4 4.662 0.000028080321 0.000084240964
## [9,] 2 3 4.072 0.000186374414 0.000372748828
## [10,] 4 5 3.834 0.000389221804 0.000389221804A continuación, averiguamos qué contrastes verifican \(\widetilde{P}_{j}\leq 0.05\):
## est.contraste.pares p.valores p.valores.ajust
## [1,] 1 4 14.296 0.000000 0.000000
## [2,] 1 5 18.130 0.000000 0.000000
## [3,] 2 5 12.568 0.000000 0.000000
## [4,] 1 3 9.634 0.000000 0.000000
## [5,] 2 4 8.734 0.000000 0.000000
## [6,] 3 5 8.496 0.000000 0.000000
## [7,] 1 2 5.562 0.000001 0.000006
## [8,] 3 4 4.662 0.000028 0.000084
## [9,] 2 3 4.072 0.000186 0.000373
## [10,] 4 5 3.834 0.000389 0.000389En este caso, también vemos que en todos los pares se verifica que el p-valor ajustado es menor que 0.05.
Concluimos lo mismo que en los contrastes realizados usando el método de Bonferroni: tenemos indicios suficientes para rechazar la igualdad de medias de aumento de masa celular del microorganismo para dos niveles cualquiera de presión de \(CO{}_2\).
Para realizar la comparación de parejas con R, tenemos que usar la misma función pairwise.t.test que usábamos en los contrastes por parejas usando el método de Bonferroni pero cambiando el parámetro p.adjust.method="holm":
##
## Pairwise comparisons using t tests with pooled SD
##
## data: X and F
##
## 0.0 0.083 0.29
## 0.083 0.000005550089963 - -
## 0.29 0.000000000011548 0.000372748827602 -
## 0.50 < 0.0000000000000002 0.000000000181263 0.000084240963982
## 0.86 < 0.0000000000000002 0.000000000000002 0.000000000330483
## 0.50
## 0.083 -
## 0.29 -
## 0.50 -
## 0.86 0.000389221804025
##
## P value adjustment method: holmR nos vuelve a mostrar una tabla donde las filas y las columnas son los niveles del factor y cuyos valores son los p-valores de los contrastes por parejas pero ajustados usando el método de Holm.
Observamos que todos los p-valores ajustados son menores que 0.05 llegando a la misma conclusión que antes.
7.2.3 Contraste de Duncan
El contraste de Duncan es otro método para ver en qué niveles hay diferencias.
Los pasos a realizar para llevarlo a cabo son los siguientes:
Se ordenan en forma ascendente las \(k\) medias muestrales.
Se considera cada par \(Y\) de medias muestrales y se calcula el valor absoluto \(D_Y\) de la diferencia entre las dos medias y el número \(p\) de medias que hay entre las dos (incluyendo las dos medias que comparamos).
Decidimos que existe diferencia entre estos dos niveles cuándo \(D_Y> SSR_p=r_p \sqrt{\frac{MS_E (n_i +n_j)}{2 n_i n_j}}\), dónde \(n_i\) y \(n_j\) son los tamaños de las subpoblaciones correspondientes a los dos niveles que comparamos y \(r_p\) es el menor rango significativo con \(N-k\) grados de libertad, que encontraréis en la tabla del test de Duncan. Si vais a http://images.google.com y escribís “tabla del test de Duncan” en la casilla de búsqueda, encontraréis un montón de tablas del test de Duncan.
La primera columna de la tabla del test de Duncan corresponde a los grados de libertad del error \(N-k\), y la primera fila, al valor \(p\). Para calcular el valor \(r_p\), tenéis que buscar el valor \(N-k\) en la primera columna y el valor \(p\) en la primera fila y ver dónde se intersecan.
Vamos a aplicar el test de Duncan a los datos del ejemplo que hemos desarrollado.
Las medias de los cinco niveles eran:
## nivel.Presión Incremento.celular
## 1 0.0 59.14
## 2 0.083 46.04
## 3 0.29 36.45
## 4 0.50 25.47
## 5 0.86 16.44Si los ordenamos de menor a mayor, obtenemos: \[ \overline{X}_{5\bullet} < \overline{X}_{4\bullet} < \overline{X}_{3\bullet} < \overline{X}_{2\bullet} < \overline{X}_{1\bullet}. \] A continuación, calculamos las medias siguientes:
- \(\overline{X}_{1\bullet}-\overline{X}_{5\bullet}\) (\(p=5\))
- \(\overline{X}_{1\bullet}-\overline{X}_{4\bullet}\) (\(p=4\))
- \(\overline{X}_{1\bullet}-\overline{X}_{3\bullet}\) (\(p=3\))
- \(\overline{X}_{1\bullet}-\overline{X}_{2\bullet}\) (\(p=2\))
- \(\overline{X}_{2\bullet}-\overline{X}_{5\bullet}\) (\(p=4\))
- \(\overline{X}_{2\bullet}-\overline{X}_{4\bullet}\) (\(p=3\))
- \(\overline{X}_{2\bullet}-\overline{X}_{3\bullet}\) (\(p=2\))
- \(\overline{X}_{3\bullet}-\overline{X}_{5\bullet}\) (\(p=3\))
- \(\overline{X}_{3\bullet}-\overline{X}_{4\bullet}\) (\(p=2\))
- \(\overline{X}_{4\bullet}-\overline{X}_{5\bullet}\) (\(p=2\))
Tenemos que \(n_i =10\) para cada \(i=1,2,3,4,5\). El valor \(SSR_p\) será, por tanto: \[ SSR_p=r_p \sqrt{\frac{MS_E}{10}} \] Calculamos los valores de \(r_p\) cada \(p=2,3,4,5\) y con el nivel de significación \(\alpha =0.05\): (usamos 40 como grados de libertad del error para consultar en la tabla, puesto que es el valor más próximo a \(N-k=45\))
| \(p\) | 2 | 3 | 4 | 5 |
|---|---|---|---|---|
| \(r_p (con\ N-k=40)\) | 2.858 | 3 | 3.102 | 3.171 |
| \(SSR_p\) | 4.76 | 4.996 | 5.166 | 5.281 |
Resumimos todo el cálculo realizado:
| Diferencias | \(d\) | \(p\) | \(SSR_p\) | \(d > SSR_p\)? | Conclusión |
|---|---|---|---|---|---|
| \(\overline{X} _{1\bullet}-\overline{X}_{5\bullet}\) | \(42.7\) | \(5\) | \(5.281\) | Sí | \(\mu_1\not=\mu_5\) |
| \(\overline{X}_{1\bullet}-\overline{X}_{4\bullet}\) | \(33.67\) | \(4\) | \(5.166\) | Sí | \(\mu_1\not=\mu_4\) |
| \(\overline{X}_{1\bullet}-\overline{X}_{3\bullet}\) | \(22.69\) | \(3\) | \(4.996\) | Sí | \(\mu_1\not=\mu_3\) |
| \(\overline{X}_{1\bullet}-\overline{X}_{2\bullet}\) | \(13.1\) | \(2\) | \(4.760\) | Sí | \(\mu_1\not=\mu_2\) |
| \(\overline{X}_{2\bullet}-\overline{X}_{5\bullet}\) | \(29.6\) | \(4\) | \(5.166\) | Sí | \(\mu_2\not=\mu_5\) |
| Diferencias | \(d\) | \(p\) | \(SSR_p\) | \(d > SSR_p\)? | Conclusión |
|---|---|---|---|---|---|
| \(\overline{X}_{2\bullet}-\overline{X}_{4\bullet}\) | \(20.57\) | \(3\) | \(4.996\) | Sí | \(\mu_2\not=\mu_4\) |
| \(\overline{X}_{2\bullet}-\overline{X}_{3\bullet}\) | \(9.59\) | \(2\) | \(4.760\) | Sí | \(\mu_2\not=\mu_3\) |
| \(\overline{X}_{3\bullet}-\overline{X}_{5\bullet}\) | \(20.01\) | \(3\) | \(4.996\) | Sí | \(\mu_3\not=\mu_5\) |
| \(\overline{X}_{3\bullet}-\overline{X}_{4\bullet}\) | \(10.98\) | \(2\) | \(4.760\) | Sí | \(\mu_3\not=\mu_4\) |
| \(\overline{X}_{4\bullet}-\overline{X}_{5\bullet}\) | \(9.03\) | \(2\) | \(4.760\) | Sí | \(\mu_4\not=\mu_5\) |
7.2.4 Contraste de Duncan en R
En R, el contraste de Duncan se realiza con la función duncan.test del paquete agricolae. La sintaxis es:
donde
aoves el resultado del ANOVA de partida,- el
factores el factor del ANOVA, grouppuede serTRUEoFALSEdependiendo de cómo queremos ver el resultado,- el
sufixesgroupsigroup=TRUEycomparisonsigroup=FALSE.
Ejemplo
Vamos a aplicar el test de Duncan a los datos de nuestro ejemplo:
##
## Attaching package: 'agricolae'## The following objects are masked from 'package:timeDate':
##
## kurtosis, skewness## The following objects are masked from 'package:EnvStats':
##
## kurtosis, skewness## difference pvalue signif. LCL UCL
## 0.0 - 0.083 13.10 0.0000 *** 8.356 17.84
## 0.0 - 0.29 22.69 0.0000 *** 17.702 27.68
## 0.0 - 0.50 33.67 0.0000 *** 28.521 38.82
## 0.0 - 0.86 42.70 0.0000 *** 37.435 47.97
## 0.083 - 0.29 9.59 0.0002 *** 4.846 14.33
## 0.083 - 0.50 20.57 0.0000 *** 15.582 25.56
## 0.083 - 0.86 29.60 0.0000 *** 24.451 34.75
## 0.29 - 0.50 10.98 0.0000 *** 6.236 15.72
## 0.29 - 0.86 20.01 0.0000 *** 15.022 25.00
## 0.50 - 0.86 9.03 0.0004 *** 4.286 13.77Nos da una tabla donde las filas son las comparaciones entre los distintos pares del niveles del factor.
La tabla contiene 5 columnas:
- la primera nos da la diferencia entre las dos medias de los dos niveles que se comparan,
- la segunda nos da el p-valor que indica si hay o no diferencias entre las dos medias,
- la tercera indica si la diferencia es significativa o no. Cuantos más asteriscos (
*) aparezcan, más significativa es la diferencia y - las dos últimas representan un intervalo de confianza para la diferencia de medias.
Si en lugar de asignar el valor FALSE al parámetro group, le asignamos el valor TRUE, obtenemos lo siguiente:
## X groups
## 0.0 59.14 a
## 0.083 46.04 b
## 0.29 36.45 c
## 0.50 25.47 d
## 0.86 16.44 eSi hubiese dos niveles donde las medias no fuesen significativamente diferentes aparecerían en la columna groups de la tabla anterior.
Por ejemplo, si las medias en los niveles 0.0 y 0.50 no fuesen significativamente diferentes, observaríamos el valor ad en la columna groups.
Como no vemos ningún caso en que aparezcan dos letras, podemos concluir que las medias de cualquier par de niveles son significativamente diferentes.
7.2.5 Contraste de Tukey
Si la tabla de datos es balanceada, es decir, todas las submuestras correspondientes a cada uno de los niveles del factor tienen el mismo tamaño, el método más preciso de comparación de medias es el llamado método de Tukey.
Es un test similar al t-test pero el estadístico de contraste tiene una distribución diferente. Podéis consultar el test en el link de la Wikipedia.
Par aplicar el contraste de Tukey en R hay que usar la función TukeyHSD (Honestly Significative Difference) y aplicarla al resultado de haber aplicado la función aov:
Ejemplo
Apliquemos el test de Tukey a los datos de nuestro ejemplo al ser la tabla de datos balanceada:
## Tukey multiple comparisons of means
## 95% family-wise confidence level
##
## Fit: aov(formula = X ~ F)
##
## $F
## diff lwr upr p adj
## 0.083-0.0 -13.10 -19.79 -6.408 0.0000
## 0.29-0.0 -22.69 -29.38 -15.998 0.0000
## 0.50-0.0 -33.67 -40.36 -26.978 0.0000
## 0.86-0.0 -42.70 -49.39 -36.008 0.0000
## 0.29-0.083 -9.59 -16.28 -2.898 0.0017
## 0.50-0.083 -20.57 -27.26 -13.878 0.0000
## 0.86-0.083 -29.60 -36.29 -22.908 0.0000
## 0.50-0.29 -10.98 -17.67 -4.288 0.0003
## 0.86-0.29 -20.01 -26.70 -13.318 0.0000
## 0.86-0.50 -9.03 -15.72 -2.338 0.0034Observando los p-valores, concluimos que hay diferencias entre todas las submuestras correspondientes a los 5 niveles de la presión.
7.3 Efectos aleatorios
En el modelo de efectos fijos que es el que hemos visto hasta ahora, el experimentador elige los niveles a estudiar.
Cuando el número de niveles es muy grande, y se quiere averiguar si los niveles del factor tienen influencia en el valor medio del parámetro con el contraste: \[ \left. \begin{array}{l} H_0 : \mbox{Las medias de todos los niveles son iguales,} \\ H_1 : \mbox{No es cierto que todos los niveles tengan la misma media.} \end{array} \right\} \] una posibilidad es elegir una m.a.s. de niveles, \(k\), y aplicar la técnica ANOVA a estos niveles.
Este es el modelo de efectos aleatorios.
Las suposiciones del modelo son:
Los \(k\) niveles elegidos forman una m.a.s. del conjunto de niveles.
Las medias \(\mu_i\) de todos los niveles siguen una distribución normal con valor medio \(\mu\) (el valor medio de toda la población) y desviación típica \(\sigma_{Tr}\).
Todas las poblaciones, para todos los niveles, siguen leyes normales.
Todas las poblaciones, para todos los niveles, tienen la misma varianza \(\sigma^2\). (homocedasticidad)
Las \(k\) muestras son m.a.s. independientes extraídas de las \(k\) poblaciones elegidas.
Una vez elegidos los \(k\) niveles, calculamos \(MS_{Tr}\) y cómo \(MS_E\) antes. Con las hipótesis anteriores, en este caso
\(E(MS_{Tr})=\displaystyle\sigma^2 + \frac{N-\sum_{i=1}^k \frac{n_i^2}{N}}{k-1}\cdot \sigma_{Tr}^2\),
\(E(MS_E)=\sigma^2\).
Si la hipótesis nula \(H_0\) es cierta, todas las medias de todos los niveles son iguales, es decir, \(\sigma_{Tr}^2=0\), y por lo tanto \(F=\frac{MS_{Tr}}{MS_E}\approx 1.\)
Si la hipótesis nula \(H_0\) es cierta, este estadístico \(F\) tiene distribución \(F_{k-1,N-k}\).
Por lo tanto, el test ANOVA es el mismo que en el caso de efectos fijos, pero usando los niveles seleccionados.
7.3.1 Condiciones del ANOVA
Recordemos que para poder realizar el contraste ANOVA se debían de cumplir las condiciones siguientes:
Las \(k\) muestras son m.a.s. independientes extraídas de poblaciones \(k\) específicas con medias \(\mu_1,\ldots,\mu_k\).
Cada una de las \(k\) poblaciones sigue una ley normal.
Todas estas poblaciones tienen la misma varianza \(\sigma^2\). (homocedasticidad)
En esta sección vamos a aprender cómo comprobar la normalidad y la igualdad de varianzas de la tabla de datos tratada.
7.3.2 Normalidad
Para verificar la normalidad de cada submuestra podemos usar todos los contrastes de normalidad aprendidos en el tema de bondad de ajuste:
- Test de Kolmogorov-Smirnov-Lilliefors.
- Test de normalidad de Anderson-Darling.
- Test de Shapiro-Wilks.
- Test omnibus de D’Agostino-Pearson.
Veamos si los datos de las submuestras para cada nivel de presión son normales usando el test de Kolmogorov-Smirnov-Lilliefors:
##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: X[F == "0.0"]
## D = 0.16, p-value = 0.6Ejemplo: condiciones ANOVA
##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: X[F == "0.083"]
## D = 0.21, p-value = 0.2##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: X[F == "0.29"]
## D = 0.22, p-value = 0.2##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: X[F == "0.50"]
## D = 0.2, p-value = 0.3##
## Lilliefors (Kolmogorov-Smirnov) normality test
##
## data: X[F == "0.86"]
## D = 0.16, p-value = 0.6Vemos que según el test de Kolmogorov-Smirnov-Lilliefors, no podemos rechazar que las 5 submuestras correspondientes a cada nivel de presión sigan la distribución normal.
Ejercicio
Comprueba la normalidad de cada una de las poblaciones anteriores usando los otros tests vistos en el tema de Bondad de Ajuste.
7.3.3 Igualdad de varianzas u homocedasticidad
Para contrastar si las \(k\) submuestras tienen las mismas varianzas, se usa el test de Bartlett.
Veamos en qué consiste.
Sean \(\mathbf{x}_i=(x_{i1},x_{i2},\ldots,x_{in_i})\), \(i=1,\ldots,k\), \(k\) muestras aleatorias simples de tamaño \(n_i\), para \(i=1,\ldots,k\) de \(k\) variables aleatorias normales \(X_i\) de varianza \(\sigma_i^2\), para \(i=1,\ldots,k\).
Nos planteamos el contraste de igualdad de varianzas: \[ \left\{ \begin{array}{l} H_0 : \sigma_1^2 =\sigma_2^2 =\cdots =\sigma_k^2, \\ H_1 : \exists i,j \mid \sigma_i^2 \not=\sigma_j^2. \end{array} \right. \]
Para realizar el contraste anterior, usaremos el estadístico de Bartlett: \[ K^2 =\frac{(N-k)\ln(\tilde{s}_p^2)-\sum\limits_{i=1}^k (n_i-1)\ln(\tilde{s}_i^2)}{1+\frac{1}{3(k-1)}\left(\sum\limits_{i=1}^k \left(\frac{1}{n_i-1}\right)-\frac{1}{N-k}\right)}, \] donde: \[ N=\sum_{i=1}^k n_i,\ \tilde{s}_p^2 =\frac{\sum\limits_{i=1}^k (n_i-1)\tilde{s}_i^2}{N-k},\ \tilde{s}_i^2 =\frac{\sum\limits_{j=1}^{n_i} (x_{ij}-\overline{x}_i)^2}{n_i-1}. \]
El estadístico anterior sigue aproximadamente la distribución \(\chi^2_{k-1}\) si la hipótesis nula es cierta.
Intuitivamente, si la hipótesis nula es cierta, o, si las varianzas son iguales (\(\sigma_1^2 =\sigma_2^2 =\cdots =\sigma_k^2\)), tendremos que \[ \tilde{s}_1^2\approx \tilde{s}_2^2\approx\cdots\approx \tilde{s}_k^2\approx \tilde{s}_p^2, \] y el valor del estadístico \(K^2\) será pequeño.
El p-valor del contraste se calcula como: \[ p=P(\chi^2_{k-1} > K^2). \]
Realizar el test de Bartlett “a mano” es bastante tedioso. Por dicho motivo, explicaremos cómo realizarlo en R.
En R hay que usar la función bartlett.test:
Ejemplo: Test de Bartlett
Apliquemos el test de Bartlett para los datos de nuestro ejemplo:
##
## Bartlett test of homogeneity of variances
##
## data: X by F
## Bartlett's K-squared = 1.1, df = 4, p-value = 0.9Como el p-valor es muy grande, concluimos que no tenemos evidencias para rechazar la igualdad de varianzas para las 5 submuestras consideradas.
7.4 Bloques completos aleatorios
El contraste ANOVA de efectos fijos o efectos aleatorios generaliza el contraste de dos medias independientes al contraste de \(k\) medias independientes.
Nos podemos preguntar si existe una generalización de contraste de dos medias dependientes a \(k\) medias dependientes.
En esta sección, veremos un contraste “ANOVA” que hace dicha generalización: el contraste de bloques completos aleatorios.
Más concretamente, supongamos que tenemos una tabla de datos como en el caso del contraste ANOVA de un factor.
O sea, queremos estudiar si las medias de una variable \(X\) segmentada en \(k\) muestras definidas por los niveles de otra variable factor \(F\) son iguales o no.
La diferencia fundamental con respecto al ANOVA de un factor es que sospechamos que hay otra variable extraña que nos puede distorsionar los resultados.
Por dicho motivo, creamos bloques a partir de dicha variable extraña para reducir su efecto. Veamos cómo.
Suponemos que tenemos \(k\) tratamientos que queremos comparar.
Escogemos como bloques conjuntos de individuos \(k\) relacionados (por ejemplo, \(k\) copias del mismo individuo).
Dentro de cada bloque, asignamos aleatoriamente a cada individuo un tratamiento.
Estos bloques vienen a ser los emparejamientos de los datos en los contrastes de medias dependientes.
En un contraste de bloques completos aleatorios,
Se han emparejado los individuos en bloques. (bloques)
Los tratamientos se asignan de manera aleatoria dentro de los bloques. (aleatorios)
Cada tratamiento se usa exactamente una vez dentro de cada bloque. (completos)
En cuanto a los tratamientos, es de efectos fijos. (la inferencia será válida sólo para los tratamientos usados)
En cuanto a los bloques, puede ser de efectos fijos (se eligen todos los bloques adecuados) o aleatorio, en este último caso el modelo es mixto.
7.4.1 Tabla de datos
Los datos se presentan en una tabla de la forma:
| Bloques/Tratamientos | Tratamiento 1 | Tratamiento 2 | \(\ldots\) | Tratamiento \(k\) |
|---|---|---|---|---|
| 1 | \(X_{11}\) | \(X_{21}\) | \(\ldots\) | \(X_{k1}\) |
| 2 | \(X_{12}\) | \(X_{22}\) | \(\ldots\) | \(X_{k2}\) |
| \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) |
| \(b\) | \(X_{1b}\) | \(X_{2b}\) | \(\ldots\) | \(X_{kb}\) |
Fijaos que ahora la fila \(j\)-ésima de la tabla de datos corresponde a los datos de la variable \(X\) para los individuos del bloque \(j\)-ésimo y la columna \(i\)-ésima, a los datos de la variable \(X\) para los individuos tratados con el tratamiento \(i\)-ésimo.
O sea, \(X_{ij}\) es el valor del tratamiento \(i\)-ésimo en el individuo correspondiente del bloque \(j\)-ésimo.
7.4.2 Contraste a realizar
El contraste que se quiere realizar es el siguiente: \[ \left. \begin{array}{l} H_0 : \mu_{1\bullet} =\mu_{2\bullet} =\cdots =\mu_{k\bullet}, \\ H_1 : \exists i,j \mid \mu_{i\bullet} \not=\mu_{j\bullet}. \end{array} \right\} \] donde cada \(\mu_{i\bullet}\) representa la media del tratamiento \(i\)-ésimo.
Ejemplo
Queremos determinar si la energía que se requiere para llevar a cabo tres actividades físicas (correr, pasear y montar en bicicleta) es la misma o no. Para cuantificar esta energía, medimos el número de Kcal. consumidas por Km. recorrido.
Las diferencias metabólicas entre los individuos pueden afectar la energía requerida para llevar a cabo una determinada actividad. Ésta sería la variable extraña que nos puede distorsionar los resultados.
Por lo tanto, no es aconsejable elegir tres grupos de individuos y a cada uno hacerle hacer una de las tres actividades físicas: las diferencias metabólicas entre los individuos elegidos podrían afectar los resultados y dar demasiada variación.
Lo que hacemos es seleccionar algunos individuos al azar (los bloques aleatorios), pedir a cada uno que corra, ande y recorra en bicicleta una distancia fijada, y determinar para cada individuo el número de Kcal. consumidas por Km. durante cada actividad.
Cada individuo es utilizado como un bloque. Las actividades se realizan en orden aleatorio, con tiempo de recuperación entre una y otra.
Al emparejar cada individuo con él mismo, eliminamos el efecto de la variación individual.
Por tanto, vamos a realizar un Diseño de bloques completos aleatorios mixto.
En la tabla siguiente se muestran los resultados obtenidos para 8 individuos:
| Bloque/Tratamiento | 1 (corriendo) | 2 (andando) | 3 (pedaleando) |
|---|---|---|---|
| 1 | 1.4 | 1.1 | 0.7 |
| 2 | 1.5 | 1.2 | 0.8 |
| 3 | 1.8 | 1.3 | 0.7 |
| 4 | 1.7 | 1.3 | 0.8 |
| 5 | 1.6 | 0.7 | 0.1 |
| 6 | 1.5 | 1.2 | 0.7 |
| 7 | 1.7 | 1.1 | 0.4 |
| 8 | 2.0 | 1.3 | 0.6 |
El contraste que queremos realizar es el siguiente: \[ \left. \begin{array}{l} H_0 : \mu_{1\bullet} = \mu_{2\bullet} = \mu_{3\bullet}, \\ H_1 : \exists i,j \mid \mu_{i\bullet}\not=\mu_{j\bullet}, \end{array} \right\} \] donde \(\mu_{i\bullet}\), \(i=1,2,3\) representa la media de Kcal. consumidas por Km. mientras se corre, se pasea o se monta en bicicleta, respectivamente.
7.4.3 Modelo
La expresión matemática del modelo a estudiar consiste ahora en expresar los datos en cuatro sumandos : la media global, las diferencias de las medias de cada nivel respecto de la media global, las diferencias de las medias de cada bloque respecto la media global y los errores residuales: \[ X_{ij} =\mu + (\mu_{i\bullet}-\mu) +(\mu_{\bullet j}-\mu) + E_{ij}, \ i=1,\ldots,k,\ j=1,\ldots,b, \]
dónde:
\(X_{ij}\): valor del tratamiento \(i\)-ésimo en el bloque \(j\)-ésimo.
\(\mu\): media global.
\(\mu_{i\bullet}\): media del tratamiento \(i\)-ésimo.
\(\mu_{\bullet j}\): media del bloque \(j\)-ésimo.
\(\mu_{i\bullet}-\mu\): efecto del tratamiento \(i\)-ésimo. (efecto tratamiento)
\(\mu_{\bullet j}-\mu\): efecto de pertenecer al bloque \(j\)-ésimo. (efecto bloque)
\(E_{ij}\): error residual o aleatorio.
7.4.4 El modelo
Las suposiciones del modelo son:
Las \(k\cdot b\) observaciones constituyen muestras aleatorias independientes, cada una de tamaño \(1\), de \(k\cdot b\) poblaciones.
Estas \(k\cdot b\) poblaciones son todas normales y con la misma varianza \(\sigma^2\).
El efecto de los bloques y los tratamientos es aditivo: no hay interacción entre los bloques y los tratamientos:
La diferencia de las medias poblacionales de cada pareja concreta de bloques es la misma para cada tratamiento.
La diferencia de las medias poblacionales de cada pareja concreta de tratamientos es la misma para cada bloque.
7.4.5 No interacción
Expliquemos con más detalle qué entendemos cuando no hay interacción entre bloques y tratamientos.
Consideremos dos ejemplos en que tenemos dos bloques: hombres y mujeres y tres tratamientos o niveles.
Las tablas siguientes nos dan las medias poblacionales de cada bloque/tratamiento dentro de cada grupo para los dos ejemplos.
En el primer caso, no tenemos interacción. En cambio, en el segundo caso, sí hay interacción.
- Caso de no interacción.
| Bloque/Tratamiento | 1 (corriendo) | 2 (andando) | 3 (pedaleando) |
|---|---|---|---|
| Hombres | \(\mu_{11}=4\) | \(\mu_{21}=5\) | \(\mu_{31}=7\) |
| Mujeres | \(\mu_{12}=3\) | \(\mu_{22}=4\) | \(\mu_{32}=6\) |
- Caso de interacción.
| Bloque/Tratamiento | 1 (corriendo) | 2 (andando) | 3 (pedaleando) |
|---|---|---|---|
| Hombres | \(\mu_{11}=4\) | \(\mu_{21}=5\) | \(\mu_{31}=7\) |
| Mujeres | \(\mu_{12}=3\) | \(\mu_{22}=4\) | \(\mu_{32}=2\) |
En el primer caso no hay interacción ya que la diferencia de las medias poblacionales de cada pareja concreta de bloques es la misma para cada tratamiento: \[ \mu_{11}-\mu_{12}=\mu_{21}-\mu_{22}=\mu_{31}-\mu_{32}=1. \] De la misma manera, la diferencia de las medias poblacionales de cada pareja concreta de tratamientos es la misma para cada bloque: \[ \begin{array}{ll} & \mu_{11}-\mu_{21}=\mu_{12}-\mu_{22}=-1,\ \mu_{21}-\mu_{31}=\mu_{22}-\mu_{32}=-2,\\ & \mu_{11}-\mu_{31}=\mu_{12}-\mu_{32}=-3. \end{array} \]
En cambio, en el segundo caso sí hay interacción ya que la diferencia de las medias poblacionales de cada pareja concreta de bloques no es la misma para cada tratamiento: \[ \mu_{11}-\mu_{12}=\mu_{21}-\mu_{22}=1\neq \mu_{31}-\mu_{32}=5. \] De la misma manera, la diferencia de las medias poblacionales de cada pareja concreta de tratamientos tampoco es la misma para cada bloque: \[ \begin{array}{ll} & \mu_{11}-\mu_{21}=\mu_{12}-\mu_{22}=-1,\ \mu_{21}-\mu_{31}=-2\neq \mu_{22}-\mu_{32}=2,\\ & \mu_{11}-\mu_{31}=-3\neq \mu_{12}-\mu_{32}=1. \end{array} \]
7.4.6 Estadísticos
Definimos los siguientes estadísticos de cara a realizar el estudio:
\(T_{i\bullet} = \sum\limits_{j=1}^b X_{ij}\), suma total del tratamiento \(i\)-ésimo, \(i=1,2,\ldots,k\).
\(\overline{X}_{i\bullet} =\dfrac{T_{i\bullet}}{b}\), media muestral del tratamiento \(i\)-ésimo, \(i=1,2,\ldots,k\).
\(T_{\bullet j}=\sum\limits_{i=1}^k X_{ij}\), suma total del bloque \(j\)-ésimo, \(j=1,2,\ldots,b\).
\(\overline{X}_{\bullet j} =\dfrac{T_{\bullet j}}{k}\), media muestral del bloque \(j\)-ésimo, \(j=1,2,\ldots,b\).
\(T_{\bullet\bullet}=\sum\limits_{i=1}^k\sum\limits_{j=1}^b X_{ij}=\sum\limits_{i=1}^k T_{i\bullet}= \sum\limits_{j=1}^b T_{\bullet j}\), suma total.
\(\overline{X}_{\bullet\bullet}=\dfrac{T_{\bullet\bullet}}{k\cdot b}\), media muestral global.
\(T^{(2)}_{\bullet\bullet}=\sum\limits_{i=1}^k\sum\limits_{j=1}^b X_{ij}^2\), suma total de cuadrados.
Ejemplo: Cuantificación Energía (continuación) Vamos a calcular los estadísticos anteriores para nuestro ejemplo de cuantificación de la energía consumida al realizar distintos ejercicios físicos.
En primer lugar, tal como hicimos con el ejemplo de ANOVA de un factor, transformamos nuestra tabla de datos en una tabla de datos de 3 columnas: en la primera habrá el número de Kcal. consumidas, en la segunda el tipo de ejercicio físico (tratamiento) y en la tercera el individuo que ha realizado el ejercicio físico (bloque):
kilocal = c(1.4,1.1,0.7,1.5,1.2,0.8,1.8,1.3,
0.7,1.7,1.3,0.8,1.6,0.7,0.1,1.5,
1.2,0.7,1.7,1.1,0.4,2.0,1.3,0.6)
tratamiento = rep(1:3,8)
bloque = rep(1:8,each=3)
tabla.datos.ANOVA.BLOQUES = data.frame(kilocal,tratamiento,bloque)## kilocal tratamiento bloque
## 1 1.4 1 1
## 2 1.1 2 1
## 3 0.7 3 1
## 4 1.5 1 2
## 5 1.2 2 2
## 6 0.8 3 2Calculemos a continuación los estadísticos definidos:
- Suma total del tratamiento \(i\)-ésimo:
(sumas.tratamientos = aggregate(kilocal ~ tratamiento,
data = tabla.datos.ANOVA.BLOQUES, FUN="sum"))## tratamiento kilocal
## 1 1 13.2
## 2 2 9.2
## 3 3 4.8- Media muestral del tratamiento \(i\)-ésimo:
(medias.tratamientos = aggregate(kilocal ~ tratamiento,
data = tabla.datos.ANOVA.BLOQUES, FUN="mean"))## tratamiento kilocal
## 1 1 1.65
## 2 2 1.15
## 3 3 0.60- Suma total del bloque \(j\)-ésimo:
## bloque kilocal
## 1 1 3.2
## 2 2 3.5
## 3 3 3.8
## 4 4 3.8
## 5 5 2.4
## 6 6 3.4
## 7 7 3.2
## 8 8 3.9- Media muestral del bloque \(j\)-ésimo:
## bloque kilocal
## 1 1 1.067
## 2 2 1.167
## 3 3 1.267
## 4 4 1.267
## 5 5 0.800
## 6 6 1.133
## 7 7 1.067
## 8 8 1.300- Suma total:
## [1] 27.2- Media muestral global:
## [1] 1.133- Suma total de cuadrados:
## [1] 36.187.4.7 Identidad de la suma de cuadrados
En el diseño de ANOVA por bloques, la variabilidad de los datos respecto la media global, lo que llamamos Suma Total de Cuadrados se descompone de tres variabilidades:
La variabilidad debida a los tratamientos.
La variabilidad debida a los bloques.
La variabilidad debida a factores aleatorios.
El siguiente teorema nos da dicha descomposición.
7.4.8 Identidad de la suma de cuadrados
donde:
\(SS_{Total} = \sum\limits_{i=1}^k\sum\limits_{j=1}^b (X_{ij}- \overline{X}_{\bullet\bullet})^2\), variabilidad total.
\(SS_{Tr}=b\sum\limits_{i=1}^k (\overline{X}_{i\bullet}-\overline{X}_{\bullet\bullet})^2\), variabilidad debida a los tratamientos.
\(SS_{Bl}=k\sum\limits_{j=1}^b (\overline{X}_{\bullet j}-\overline{X}_{\bullet\bullet})^2\), variabilidad debida a los bloques.
7.4.9 Identidad de la suma de cuadrados
- \(SS_E= \sum\limits_{i=1}^k\sum\limits_{j=1}^b (X_{ij} - \overline{X}_{i\bullet}- \overline{X}_{\bullet j}+\overline{X}_{\bullet\bullet})^2\), variabilidad debida a factores aleatorios.
Usando la notación definida, el teorema anterior puede escribirse de la forma siguiente:
\[ SS_{Total}=SS_{Tr}+SS_{Bl}+SS_E. \]
Para calcular las variabilidades definidas anteriormente, hay que usar las expresiones siguientes:
\(\displaystyle SS_{Total}=T_{\bullet\bullet}^{(2)}-\dfrac{T_{\bullet\bullet}^2}{k\cdot b}\).
\(\displaystyle SS_{Tr}= \sum\limits_{i=1}^k\dfrac{T_{i\bullet}^2}{b}-\dfrac{T_{\bullet\bullet}^2}{k\cdot b}\).
\(\displaystyle SS_{Bl}= \sum\limits_{j=1}^b \dfrac{T_{\bullet j}^2}{k}-\dfrac{T_{\bullet\bullet}^2}{k\cdot b}\).
\(SS_E=SS_{Total}-SS_{Tr}-SS_{Bl}\).
Calculemos las variabilidades definidas para los datos del ejemplo que estamos desarrollando:
- Variabilidad total:
## [1] 5.353- Variabilidad debida a los tratamientos:
num.bloques = 8
num.tratamientos = 3
(SS.Tr = (1/num.bloques)*sum(sumas.tratamientos[,2]^2)-
suma.total^2/nrow(tabla.datos.ANOVA.BLOQUES))## [1] 4.413- Variabilidad debida a los bloques:
(SS.Bl = (1/num.tratamientos)*sum(sumas.bloques[,2]^2)-
suma.total^2/nrow(tabla.datos.ANOVA.BLOQUES))## [1] 0.5533- Variabilidad debida a factores aleatorios:
## [1] 0.38677.4.10 Contraste
Recordemos que el contraste a realizar es el siguiente:
\[ \left. \begin{array}{l} H_0 : \mu_{1\bullet}=\cdots =\mu_{k\bullet}, \\ H_1 : \exists i,j=1,\ldots ,k\mid \mu_{i\bullet} \neq \mu_{j\bullet}. \end{array} \right\} \] Para realizar dicho contraste, usaremos los estadísticos siguientes:
Cuadrado medio de los tratamientos: \(MS_{Tr}=\dfrac{SS_{Tr}}{k-1}\).
Cuadrado medio del error: \(MS_E = \dfrac{SS_E}{(b-1) (k-1)}\).
Cuadrado medio de los bloques: \(MS_{Bl}=\dfrac{SS_{Bl}}{b-1}\).
7.4.11 Contraste
El valor medio o la esperanza de los estadísticos \(MS_{Tr}\) y \(MS_E\) es el siguiente:
\[ \begin{array}{l} E(MS_{Tr})=\sigma^2 + \dfrac{b}{k-1}\sum\limits_{i=1}^k (\mu_{i\bullet}-\mu)^2, \\ E(MS_E)=\sigma^2. \end{array} \]
Entonces, si \(H_0:\mu_{1\bullet}=\cdots =\mu_{k\bullet}=\mu\) es cierta, se verificará que la cantidad siguiente será nula:
\[ \sum\limits_{i=1}^k (\mu_{i\bullet}-\mu)^2 = 0, \] y si \(H_0\) no es cierta, dicha cantidad sería positiva.
7.4.12 Estadísticos del contraste
Basándonos en la apreciación anterior, se considera como estadístico de contraste el cociente siguiente:
\[ F=\frac{MS_{Tr}}{MS_E}, \] que, si \(H_0\) es cierta, se distribuye según una \(F_{k-1,(k-1)(b-1)}\) (F de Fisher con \(k-1\) y grados \((k-1)(b-1)\) de libertad).
Por tanto, su valor será próximo a \(1\).
Entonces, rechazaremos la hipótesis nula si \(F\) es bastante más grande que 1 basándonos en el p-valor:
\[ p=P(F_{k-1,(k-1)(b-1)}\geq F), \] con el significado usual: si el p-valor es más pequeño que el nivel de significación \(\alpha\), rechazamos \(H_0\) y concluimos que no todas las medias son iguales. En caso contrario, aceptamos \(H_0\).
Realicemos el contraste ANOVA por bloques en el ejemplo que vamos desarrollando.
Los cuadrados medios serán:
- Cuadrado medio de los tratamientos, \(MS_{Tr}=\dfrac{SS_{Tr}}{k-1}\):
## [1] 2.207- Cuadrado medio del error, \(MS_E = \dfrac{SS_E}{(b-1) (k-1)}\):
## [1] 0.02762- Cuadrado medio de los bloques, \(MS_{Bl}=\dfrac{SS_{Bl}}{b-1}\):
## [1] 0.07905- Estadístico de contraste, \(F=\frac{MS_{Tr}}{MS_E}\):
## [1] 79.9- p-valor, \(p=P(F_{k-1,(k-1)(b-1)}\geq F)\):
## [1] 0.00000002201Como el p-valor es muy pequeño, concluimos que tenemos indicios suficientes para rechazar que las medias de los tratamientos sean iguales. Es decir, la energía consumida al correr, pasear y montar en bicicleta no es la misma para los tres casos.
7.4.13 Tabla del contraste
El contraste realizado se resume en la tabla siguiente:
| Origen variación | Grados de libertad | Sumas de cuadrados | Cuadrados medios | Estadístico de contraste | p-valor |
|---|---|---|---|---|---|
| Tratamiento | \(k-1\) | \(SS_{Tr}\) | \(MS_{Tr}=\frac{SS_{Tr}}{k-1}\) | \(F=\frac{MS_{Tr}}{MS_E}\) | p-valor |
| Bloque | \(b-1\) | \(SS_{Bl}\) | \(MS_{Bl}=\dfrac{SS_{Bl}}{b-1}\) | ||
| Error | \((k-1)\cdot (b-1)\) | \(SS_E\) | \(MS_E=\frac{SS_E}{(k-1)\cdot (b-1)}\) |
En el ejemplo que estamos estudiando, la tabla ANOVA por bloques es la siguiente:
| Origen variación | Grados de libertad | Sumas de cuadrados | Cuadrados medios | Estadístico de contraste | p-valor |
|---|---|---|---|---|---|
| Tratamiento | \(2\) | \(4.413\) | \(2.207\) | \(79.897\) | 0 |
| Bloque | \(7\) | \(0.553\) | \(0.079\) | ||
| Error | \(14\) | \(0.387\) | \(0.028\) |
7.5 ANOVA por bloques en R
Para realizar un contraste ANOVA por bloques en R, hay que usar la misma función aov que hemos usado cuando explicamos el ANOVA de un factor.
Recordemos que la función aov se aplica a la tabla de datos modificada que hemos explicado:
donde recordemos que en la variable X se almacenan los valores de la variable a estudiar, la variable factor Tr nos dice el tratamiento que se aplica y la variable factor Bl, el bloque correspondiente.
Ejemplo: ANOVA por bloques
El contraste ANOVA por bloques en R para nuestro ejemplo se realizaría de la forma siguiente:
## Df Sum Sq Mean Sq F value Pr(>F)
## tratamiento 1 4.41 4.41 98.31 0.0000000022 ***
## bloque 1 0.00 0.00 0.03 0.87
## Residuals 21 0.94 0.04
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Fijaos que la tabla obtenida no sería correcta ya que los grados de libertad de los tratamientos y los grados de libertad de los bloques (columna Df) son 1 y deberían ser 2 y 7, respectivamente.
Éste es uno de los errores más comunes que pueden ocurrir cuando realizamos un contraste ANOVA en R.
La razón es que R no ha considerado como factor ni la columna de los tratamientos ni la columna de los bloques en la tabla de datos tabla.datos.ANOVA.BLOQUES.
Para solucionar este problema, le decimos a R que dichas columnas son factores:
summary(aov(kilocal ~ as.factor(tratamiento) + as.factor(bloque),
data = tabla.datos.ANOVA.BLOQUES))## Df Sum Sq Mean Sq F value Pr(>F)
## as.factor(tratamiento) 2 4.41 2.207 79.90 0.000000022 ***
## as.factor(bloque) 7 0.55 0.079 2.86 0.045 *
## Residuals 14 0.39 0.028
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Observamos que hemos obtenido la misma tabla que la tabla obtenida haciendo los cálculos “a mano”.
Notemos que no nos interesa ni el segundo estadístico de contraste ni el segundo p-valor, puesto que queremos evaluar si los tratamientos tienen o no la misma efectividad (pero R no sabe quien es el bloque ni quien es el tratamiento).
7.5.1 Efectividad en la construcción de los bloques
Recordemos que el objetivo del diseño por bloques es la reducción de algunas variables extrañas que podrían distorsionarnos los resultados si hubiésemos realizado el contraste ANOVA de un factor sin tener en cuenta los bloques.
Llegados a este punto, nos podemos preguntar si el diseño por bloques ha sido efectivo, es decir, si dicha construcción realmente ha reducido el efecto de las variables extrañas que no controlamos.
Expresado en términos de la variabilidad, la efectividad del diseño por bloques significa que la variabilidad debida a los bloques, \(SS_{Bl}\), explica una parte importante de la variabilidad total, \(SST\).
En este caso, el valor de \(SSE\) disminuiría aumentando el valor del estadístico de contraste \(F\), lo que haría más “difícil” aceptar la hipótesis nula, mejorando la potencia del contraste.
La efectividad en la construcción de los bloques se estima con la eficiencia relativa, \(RE\).
Se interpreta cómo la relación entre el número de observaciones de un experimento completamente aleatorio (CA) y el número de observaciones de un experimento de bloques completo aleatorio (BCA) necesaria para obtener tests equivalentes.
Por ejemplo, si \(RE=3\) significa que el diseño CA requiere tres veces tantas observaciones cómo el diseño de BCA. En este caso, ha merecido la pena el uso de bloques.
En cambio, un valor de \(RE=0.5\) significa que con un diseño CA hubiera bastado la mitad de observaciones que al diseño BCA. En este caso, no era aconsejable el uso de bloques.
Para la estimación de la eficiencia relativa \(RE\), se usa el estadístico siguiente: \[ \widehat{RE}=c+(1-c)\frac{MS_{Bl}}{MS_E}, \] dónde \(c=\dfrac{b(k-1)}{(bk-1)}\).
Por convenio, si \(\widehat{RE}>1.25\), se entiende que la construcción de los bloques ha sido efectiva.
7.6 ANOVA de dos vías
En la sección ANOVA de un factor, realizábamos un contraste de \(k\) medias (\(k\geq 3\)) para \(k\) poblaciones o subpoblaciones clasificadas según un factor.
Cada nivel del factor, daba lugar a una subpoblación.
En esta sección, supondremos que tendremos dos factores que nos clasifican los valores de la población en las correspondientes subpoblaciones.
Como tenemos dos factores, hay tres cuestiones sobre la mesa que habrá que resolver:
- ¿Existen diferencias entre las medias de las subpoblaciones debido al factor 1?
- ¿Existen diferencias entre las medias de las subpoblaciones debido al factor 2?
- ¿Existe interacción entre los dos factores?
Concretamente, para llevar a cabo el ANOVA de dos vías, consideraremos el caso más sencillo: diseño completamente aleatorio con efectos fijos:
Usaremos dos factores (dos vías).
Usaremos todos los niveles de cada factor (efectos fijos).
Tomaremos muestras aleatorias independientes del mismo tamaño de cada combinación de niveles de los dos factores (completamente aleatorio y balanceado).
Llamaremos \(A\) y \(B\) a los factores a partir de los cuales vamos a segregar los datos de la variable cuyas medias de las subpoblaciones segregadas queremos comparar.
Supondremos que el factor \(A\) tiene \(a\) niveles y el factor \(B\), \(b\) niveles.
Tomamos \(n\) observaciones para cada combinación de tratamientos. (estudio balanceado)
Por tanto, el número total de observaciones será \(n\cdot a\cdot b\).
La variable aleatoria \(X_{ijk}\), \(i=1,\ldots,a\), \(j=1,\ldots,b\), \(k=1,\ldots,n\), nos da la respuesta de la \(k\)-ésima unidad experimental al nivel \(i\)-ésimo del factor \(A\) y el nivel \(j\)-ésimo del factor \(B\).
La tabla de los datos tendrá la estructura siguiente:
| Factor \(B\)/Factor \(A\) | \(1\) | \(2\) | \(\cdots\) | \(a\) |
|---|---|---|---|---|
| \(1\) | \(X_{111}\) | \(X_{211}\) | \(\cdots\) | \(X_{a11}\) |
| \(X_{112}\) | \(X_{212}\) | \(\cdots\) | \(X_{a12}\) | |
| \(\cdots\) | \(\cdots\) | \(\cdots\) | \(\cdots\) | |
| \(X_{11n}\) | \(X_{21n}\) | \(\cdots\) | \(X_{a1n}\) |
| Factor \(B\)/Factor \(A\) | \(1\) | \(2\) | \(\cdots\) | \(a\) |
|---|---|---|---|---|
| \(2\) | \(X_{121}\) | \(X_{221}\) | \(\cdots\) | \(X_{a21}\) |
| \(X_{122}\) | \(X_{222}\) | \(\cdots\) | \(X_{a22}\) | |
| \(\cdots\) | \(\cdots\) | \(\cdots\) | \(\cdots\) | |
| \(X_{12n}\) | \(X_{22n}\) | \(\cdots\) | \(X_{a2n}\) |
| Factor \(B\)/Factor \(A\) | \(1\) | \(2\) | \(\cdots\) | \(a\) |
|---|---|---|---|---|
| \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) | \(\vdots\) |
| \(b\) | \(X_{1b1}\) | \(X_{2b1}\) | \(\cdots\) | \(X_{ab1}\) |
| \(X_{1b2}\) | \(X_{2b2}\) | \(\cdots\) | \(X_{ab2}\) | |
| \(\cdots\) | \(\cdots\) | \(\cdots\) | \(\cdots\) | |
| \(X_{1bn}\) | \(X_{2bn}\) | \(\cdots\) | \(X_{abn}\) |
Ejemplo
En un experimento para determinar el efecto de la luz y la temperatura sobre el índice gonadosomático (GSI, una medida de crecimiento del ovario) de una especie de pez, se utilizaron dos fotoperiodos (14 horas de luz/10 horas de oscuridad, y 9 horas de luz/15 horas de oscuridad) y dos temperaturas (16\({}^\circ\) y 27 \({}^\circ\)C).
El experimento se realizó sobre \(20\) hembras. Se dividieron aleatoriamente en \(4\) subgrupos de \(5\) ejemplares cada uno. Cada grupo recibió una combinación diferente de luz y temperatura.
A los 3 meses se midieron los GSI de los peces y se obtuvieron los resultados siguientes:
7.6.1 Ejemplo
| Factor \(B\) (temperatura)/Factor \(A\) | \(9\) horas | \(14\) horas |
|---|---|---|
| \(27^\circ\)C | \(0.90\) | \(0.83\) |
| \(1.06\) | \(0.67\) | |
| \(0.98\) | \(0.57\) | |
| \(1.29\) | \(0.47\) | |
| \(1.12\) | \(0.66\) | |
| \(16^\circ\)C | \(1.30\) | \(1.01\) |
| \(2.88\) | \(1.52\) | |
| \(2.42\) | \(1.02\) | |
| \(2.66\) | \(1.32\) | |
| \(2.94\) | \(1.63\) |
7.6.2 Almacenamiento de datos en ANOVA de dos vías
Vamos a almacenar los datos de una forma parecida a la usada en ANOVA de un factor.
Sea \(X\) la variable característica de la que comparamos las medias de las subpoblaciones. Sean \(A\) y \(B\) los factores.
Vamos a transformar la tabla anterior de los datos en una tabla de datos con \(N=n\cdot a\cdot b\) filas y tres columnas.
La primera columna serán los valores de la variable \(X\), la segunda los valores o niveles de la variable factor \(A\) y la tercera, los valores o niveles de la variable factor \(B\).
La transformación de la tabla de datos para el ejemplo anterior se realizaría de la forma siguiente:
GSI = c(0.90,0.83,1.06,0.67,0.98,0.57,1.29,0.47,1.12,0.66,
1.30,1.01,2.88,1.52,2.42,1.02,2.66,1.32,2.94,1.63)
temperatura = factor(rep(c(27,16),each=10))
fotoperiodos = factor(rep(c(9,14),times=10))
tabla.datos.GSI = data.frame(GSI,temperatura,fotoperiodos)
head(tabla.datos.GSI)## GSI temperatura fotoperiodos
## 1 0.90 27 9
## 2 0.83 27 14
## 3 1.06 27 9
## 4 0.67 27 14
## 5 0.98 27 9
## 6 0.57 27 147.6.3 El modelo
Para poder realizar un ANOVA de dos factores, supondremos que los datos verifican las suposiciones siguientes:
Las observaciones para cada combinación de niveles constituyen muestras aleatorias simples independientes, cada una de tamaño \(n\), de poblaciones \(a\cdot b\),
Cada una de las \(a\cdot b\) poblaciones es normal.
Todas las \(a\cdot b\) poblaciones tienen la misma varianza, \(\sigma^2\).
Los parámetros que intervendrán en el contraste son:
\(\mu\): media poblacional global.
\(\mu_{i\bullet\bullet}\): media poblacional del nivel \(i\)-ésimo del factor \(A\).
\(\mu_{\bullet j\bullet}\): media poblacional del nivel \(j\)-ésimo del factor \(B\).
\(\mu_{ij\bullet}\): media poblacional de la combinación \((i,j)\) de niveles \(A\) de y \(B\).
En este caso la expresión matemática del modelo consiste en separar los valores de la variable \(X\) en 5 sumandos: \[ \begin{array}{l} X_{ijk} = \mu + \alpha_i + \beta_j + (\alpha\beta)_{ij}+E_{ijk},\\ \qquad\qquad i=1,\ldots,a,\ j=1,\ldots,b,\ k=1,\ldots,n \end{array} \] donde
\(\mu\): es la media global,
\(\alpha_i =\mu_{i\bullet\bullet}-\mu\): efecto al pertenecer al nivel \(i\)-ésimo del factor \(A\),
\(\beta_j =\mu_{\bullet j\bullet}-\mu\): efecto al pertenecer al nivel \(j\)-ésimo del factor \(B\),
\((\alpha\beta)_{ij}=\mu_{ij\bullet}-\mu_{i\bullet\bullet}-\mu_{\bullet j\bullet}+\mu\): efecto de la interacción entre el nivel \(i\)-ésimo del factor \(A\) y el nivel \(j\)-ésimo del factor \(B\),
\(E_{ijk}=X_{ijk}-\mu_{ij\bullet}\): error residual o aleatorio.
7.6.4 Sumas y medias
Definimos los estadísticos siguientes:
Suma y media de los datos para la combinación de niveles \(i\) y \(j\): \[T_{ij\bullet}=\sum\limits_{k=1}^n X_{ijk}, \quad \overline{X}_{ij\bullet}=\dfrac{T_{ij\bullet}}{n}.\]
Suma y media de los datos para el nivel \(i\)-ésimo: \[T_{i\bullet\bullet}=\sum\limits_{j=1}^b\sum\limits_{k=1}^n X_{ijk}=\sum\limits_{j=1}^b T_{ij\bullet},\quad \overline{X}_{i\bullet\bullet}=\dfrac{T_{i\bullet\bullet}}{bn}.\]
Suma y media de los datos para el nivel \(j\)-ésimo: \[T_{\bullet j\bullet}=\sum\limits_{i=1}^a\sum\limits_{k=1}^n X_{ijk}=\sum\limits_{i=1}^a T_{ij\bullet}, \quad \overline{X}_{\bullet j\bullet}=\dfrac{T_{\bullet j\bullet}}{an}.\]
Suma total de los datos: \[T_{\bullet\bullet\bullet}=\sum\limits_{i=1}^{a}\sum\limits_{j=1}^b\sum\limits_{k=1}^n X_{ijk}=\sum\limits_{i=1}^{a} T_{i\bullet\bullet}=\sum\limits_{j=1}^b T_{\bullet j\bullet}.\]
Media muestral de todos los datos: \[\overline{X}_{\bullet\bullet\bullet}=\dfrac{T_{\bullet\bullet\bullet}}{a b n}.\]
Suma de los cuadrados de los datos: \[T^{(2)}_{\bullet\bullet\bullet}=\sum\limits_{i=1}^{a}\sum\limits_{j=1}^b\sum\limits_{k=1}^n X_{ijk}^2.\]
Los valores de los estadísticos anteriores para los datos de nuestro ejemplo son los siguientes:
- Suma de los datos para la combinación de niveles \(i\) y \(j\):
(suma.combinación.niveles = aggregate(GSI ~ temperatura+fotoperiodos,
data = tabla.datos.GSI, FUN="sum"))## temperatura fotoperiodos GSI
## 1 16 9 12.20
## 2 27 9 5.35
## 3 16 14 6.50
## 4 27 14 3.20Los valores de los estadísticos anteriores para los datos de nuestro ejemplo son los siguientes:
- Media de los datos para la combinación de niveles \(i\) y \(j\):
(media.combinación.niveles = aggregate(GSI ~ temperatura+fotoperiodos,
data=tabla.datos.GSI, FUN="mean"))## temperatura fotoperiodos GSI
## 1 16 9 2.44
## 2 27 9 1.07
## 3 16 14 1.30
## 4 27 14 0.64- Suma y media de los datos para el nivel \(i\)-ésimo:
## fotoperiodos GSI
## 1 9 17.55
## 2 14 9.70## fotoperiodos GSI
## 1 9 1.755
## 2 14 0.970- Suma y media de los datos para el nivel \(j\)-ésimo:
## temperatura GSI
## 1 16 18.70
## 2 27 8.55## temperatura GSI
## 1 16 1.870
## 2 27 0.855- Suma total de los datos:
## [1] 27.25- Media muestral de todos los datos:
## [1] 1.363- Suma de los cuadrados de los datos:
## [1] 48.267.6.5 Identidades de sumas de cuadrados
En el caso de ANOVA de dos factores, la variabilidad total de los datos respecto la media global, (Suma Total de Cuadrados) se separa en 4 variabilidades:
la variabilidad de las medias de cada grupo del factor \(A\) respecto la media global,
la variabilidad de las medias de cada grupo del factor \(B\) respecto la media global,
la variabilidad de las medias de cada combinación de grupos de los factores \(A\) y \(B\) respecto la media global,
la variabilidad debida a factores aleatorios.
Para estimar dichas variabilidades, se introducen las sumas de cuadrados siguientes:
Estimación de la variabilidad total de los datos respecto la media global: \(SS_{Total} =\sum\limits_{i=1}^a\sum\limits_{j=1}^b\sum\limits_{k=1}^n (X_{ijk}-\overline{X}_{\bullet\bullet\bullet})^2\).
Estimación de la variabilidad de las medias de cada grupo del factor \(A\) respecto la media global: \(SS_A =b n\sum\limits_{i=1}^a (\overline{X}_{i\bullet\bullet}-\overline{X}_{\bullet\bullet\bullet})^2\).
Estimación de la variabilidad de las medias de cada grupo del factor \(B\) respecto la media global: \(SS_B =a n\sum\limits_{j=1}^b (\overline{X}_{\bullet j\bullet}-\overline{X}_{\bullet\bullet\bullet})^2\).
Estimación de la variabilidad de las medias de cada combinación de grupos de los factores \(A\) y \(B\) respecto la media global o variabilidad debida a la interacción de los factores \(A\) y \(B\): \(SS_{AB}=n \sum\limits_{i=1}^a\sum\limits_{j=1}^b (\overline{X}_{ij\bullet}-\overline{X}_{i\bullet\bullet}-\overline{X}_{\bullet j\bullet}+\overline{X}_{\bullet\bullet\bullet})^2\).
Estimación de la variabilidad que tendríamos si consideramos la combinación de factores \(A\) y \(B\) como si fuese un sólo factor: \(SS_{Tr}=n\sum\limits_{i=1}^a\sum\limits_{j=1}^b (\overline{X}_{ij\bullet}-\overline{X}_{\bullet\bullet\bullet})^2\).
Estimación de la la variabilidad debida a factores aleatorios: \(SS_E = \sum\limits_{i=1}^a\sum\limits_{j=1}^b\sum\limits_{k=1}^n (X_{ijk}-\overline{X}_{ij\bullet})^2\).
El teorema siguiente nos da la descomposición que comentamos antes:
7.6.6 Cálculo de las sumas de cuadrados
Para calcular las variabilidades, se usan las fórmulas equivalentes siguientes:
\(SS_{Total} = T_{\bullet\bullet\bullet}^{(2)} -\dfrac{T_{\bullet\bullet\bullet}^2}{abn}\).
\(SS_A = \displaystyle \dfrac1{bn}\sum\limits_{i=1}^a T_{i\bullet\bullet}^2-\dfrac{T_{\bullet\bullet\bullet}^2}{abn}\).
\(SS_B =\displaystyle\dfrac1{an}\sum\limits_{j=1}^b T_{\bullet j\bullet}^2-\dfrac{T_{\bullet\bullet\bullet}^2}{abn}\).
\(SS_{Tr}=\displaystyle\dfrac1{n}\sum\limits_{i=1}^a\sum\limits_{j=1}^b T_{ij\bullet}^2 - \dfrac{T_{\bullet\bullet\bullet}^2}{abn}\).
\(SS_{AB} = SS_{Tr} -SS_A-SS_B\).
\(SS_E = SS_{Total}-SS_{Tr}\).
Calculemos las variabilidades para los datos de nuestro ejemplo:
- Variabilidad total de los datos respecto la media global:
## [1] 11.13- Variabilidad de las medias de cada grupo del factor \(A\) respecto la media global:
## [1] 3.081- Variabilidad de las medias de cada grupo del factor \(B\) respecto la media global:
## [1] 5.151- Variabilidad que tendríamos si consideramos la combinación de factores \(A\) y \(B\) como si fuese un sólo factor:
## [1] 8.862- Variabilidad debida a la interacción de los factores \(A\) y \(B\):
## [1] 0.6301- Variabilidad debida a factores aleatorios:
## [1] 2.2717.6.7 Cuadrados medios
Para realizar el ANOVA de dos factores, usaremos los siguientes cuadrados medios:
Cuadrado medio del factor \(A\): \(MS_A =\dfrac{SS_A}{a-1}\).
Cuadrado medio del factor \(B\): \(MS_B =\dfrac{SS_B}{b-1}\).
Cuadrado medio de la interacción entre los factores \(A\) y \(B\): \(MS_{AB}=\dfrac{SS_{AB}}{(a-1)(b-1)}\).
7.6.8 Cuadrados medios
Cuadrado medio de los tratamientos: \(MS_{Tr}=\dfrac{SS_{Tr}}{ab-1}\).
Cuadrado medio residual: \(MS_E=\dfrac{SS_E}{ab (n-1)}\).
Calculemos los cuadrados medios para los datos del ejemplo que vamos desarrollando:
- Cuadrado medio del factor \(A\) (fotoperíodo):
## [1] 3.081- Cuadrado medio del factor \(B\) (temperatura):
## [1] 5.151- Cuadrado medio de la interacción entre los factores \(A\) y \(B\):
## [1] 0.6301- Cuadrado medio de los tratamientos:
## [1] 2.954- Cuadrado medio residual:
## [1] 0.1427.6.9 Contrastes a realizar
En una ANOVA de dos vías, nos pueden interesar los cuatro contrastes siguientes:
- Contraste de medias del factor \(A\): contrastamos si hay diferencias entre los niveles del factor \(A\): \[ \left\{ \begin{array}{l} H_0 : \mu_{1\bullet\bullet}=\mu_{2\bullet\bullet}=\cdots =\mu_{a\bullet\bullet}, \\ H_1 : \exists i,i'\mid \mu_{i\bullet\bullet} \not = \mu_{i'\bullet\bullet}. \end{array} \right. \]
El estadístico de contraste es \(F=\frac{MS_A}{MS_E},\) que, si la hipótesis nula \(H_0\) es cierta, tiene una distribución \(F\) de Fisher con \(a-1\) y grados \(ab(n-1)\) de libertad y valor próximo a 1.
- Contraste de medias del factor \(B\): contrastamos si hay diferencias entre los niveles del factor \(B\): \[ \left\{ \begin{array}{l} H_0 : \mu_{\bullet 1\bullet}=\mu_{\bullet 2\bullet}=\cdots =\mu_{ \bullet b\bullet}, \\ H_1 : \exists j,j'\mid \mu_{\bullet j\bullet} \not = \mu_{\bullet j'\bullet}. \end{array} \right. \]
El estadístico de contraste es \(F=\frac{MS_B}{MS_E},\) que, si la hipótesis nula \(H_0\) es cierta, tiene una distribución \(F\) de Fisher con \(b-1\) y grados \(ab(n-1)\) de libertad y valor próximo a 1.
- Contraste de los tratamientos: contrastamos si hay diferencias entre las parejas (nivel \(i\) de \(A\), nivel \(j\) de \(B\)): \[ \left\{ \begin{array}{l} H_0 : \forall i,j,i',j'\mid \mu_{ij\bullet}=\mu_{i'j'\bullet}, \\ H_1 : \exists i,j,i',j'\mid \mu_{ij\bullet}\neq \mu_{i'j'\bullet}. \end{array} \right. \]
El estadístico de contraste es \(F=\frac{MS_{Tr}}{MS_E},\) que, si la hipótesis nula \(H_0\) es cierta, tiene una distribución \(F\) de Fisher con \(ab-1\) y grados \(ab(n-1)\) de libertad y valor próximo a 1.
- Contraste de no interacción: contrastamos si hay interacción entre los factores \(A\) y \(B\) \[ \left\{ \begin{array}{l} H_0 : \forall i,j\mid (\alpha\beta)_{ij} =0, \\ H_1 : \exists i,j\mid (\alpha\beta)_{ij}. \not = 0 \end{array} \right. \]
El estadístico de contraste es \(F = \frac{MS_{AB}}{MS_E},\) que, si la hipótesis nula \(H_0\) es cierta, tiene distribución \(F\) de Fisher con \((a-1)(b-1)\) y grados \(ab(n-1)\) de libertad y valor próximo a 1.
En los cuatro casos, el p-valor es \[ P(F_{x,y}\geq \mbox{valor del estadístico}), \] donde \(F_{x,y}\) representa la distribución \(F\) de Fisher con los grados de libertad que correspondan:
- Contraste de medias del factor \(A\): \(x=a-1\), \(y=ab(n-1)\).
- Contraste de medias del factor \(B\): \(x=b-1\), \(y=ab(n-1)\).
- Contraste de los tratamientos: \(x=ab-1\), \(y=ab(n-1)\).
- Contraste de no interacción: \(x=(a-1)(b-1)\), \(y=ab(n-1)\).
Los contrastes anteriores se resumen en la tabla siguiente:
7.6.10 Tabla ANOVA
| Variación | Grados de libertad | Suma de cuadrados | Cuadrados medios | Estadístico \(F\) | p-valores |
|---|---|---|---|---|---|
| Tratamientos | \(ab-1\) | \(SS_{Tr}\) | \(MS_{Tr}\) | \(\dfrac{MS_{Tr}}{MS_E}\) | p-valor |
| \(A\) | \(a-1\) | \(SS_A\) | \(MS_{A}\) | \(\dfrac{MS_{A}}{MS_E}\) | p-valor |
| \(B\) | \(b-1\) | \(SS_B\) | \(MS_{B}\) | \(\dfrac{MS_{B}}{MS_E}\) | p-valor |
| \(AB\) | \((a-1)(b-1)\) | \(SS_{AB}\) | \(MS_{AB}\) | \(\dfrac{MS_{AB}}{MS_E}\) | p-valor |
| Error | \(ab(n-1)\) | \(SS_E\) | \(MS_{E}\) |
La tabla ANOVA para los datos de nuestro ejemplo es la siguiente:
| Variación | Grados de libertad | Suma de cuadrados | Cuadrados medios | Estadístico \(F\) | p-valores |
|---|---|---|---|---|---|
| Tratamientos | 3 | 8.862 | 2.954 | \(\dfrac{2.954}{0.142}=20.809\) | \(P(F_{3,16} > 20.809)=0\) |
| \(A\) | 1 | 3.081 | 3.081 | \(\dfrac{3.081}{0.142}=21.704\) | \(P(F_{1,16}>21.704)=0\) |
| \(B\) | 1 | 5.151 | 5.151 | \(\dfrac{5.151}{0.142}=36.285\) | \(P(F_{1,16}>36.285)=0\) |
| \(AB\) | 1 | 0.63 | 0.63 | \(\dfrac{0.63}{0.142}=4.439\) | \(P(F_{1,16}>4.439)=0.051\) |
| Error | 16 | 2.271 | 0.142 |
Las conclusiones que se obtienen de los 4 contrastes a partir de la tabla anterior son las siguientes:
- Contraste de medias del factor \(A\): como el p-valor es prácticamente 0, concluimos que tenemos indicios suficientes para rechazar que no hay diferencias entre los niveles del factor \(A\). Es decir, el índice GSI se ve afectado por el fotoperíodo.
- Contraste de medias del factor \(B\): como el p-valor es prácticamente 0, concluimos también que tenemos indicios suficientes para rechazar que no hay diferencias entre los niveles del factor \(B\). Es decir, el índice GSI se ve afectado por la temperatura.
- Contraste de los tratamientos: como el p-valor es prácticamente 0, concluimos también que tenemos indicios suficientes para rechazar que no hay diferencias entre los niveles de la combinación de factores \(A\) y \(B\). Es decir, el índice GSI se ve afectado por los cuatro niveles de la combinación fotoperíodo/temperatura.
- Contraste de no interacción: el p-valor está entre 0.05 y 0.1. Por tanto, al estar en la zona de penumbra, no podemos concluir si hay o no interacción entre los factores \(A\) y \(B\). Dicho de otra manera, no queda claro si hay interacción entre el fotoperíodo y la temperatura.
7.6.11 Contraste ANOVA de dos vías en R
Para realizar un contraste ANOVA de dos vías en R hay que usar la función aov que usábamos en los casos de ANOVA de un factor y ANOVA por bloques.
Dicha función se aplica a la tabla de datos modificada que hemos explicado:
donde X es la variable donde se almacenan los valores \(X_{ijk}\) y en las variables factor A y B, los niveles de los factores A y B, respectivamente.
Hagamos un boxplot de la variable GSI según el fotoperíodo y según la temperatura para observar gráficamente si hay diferencias:
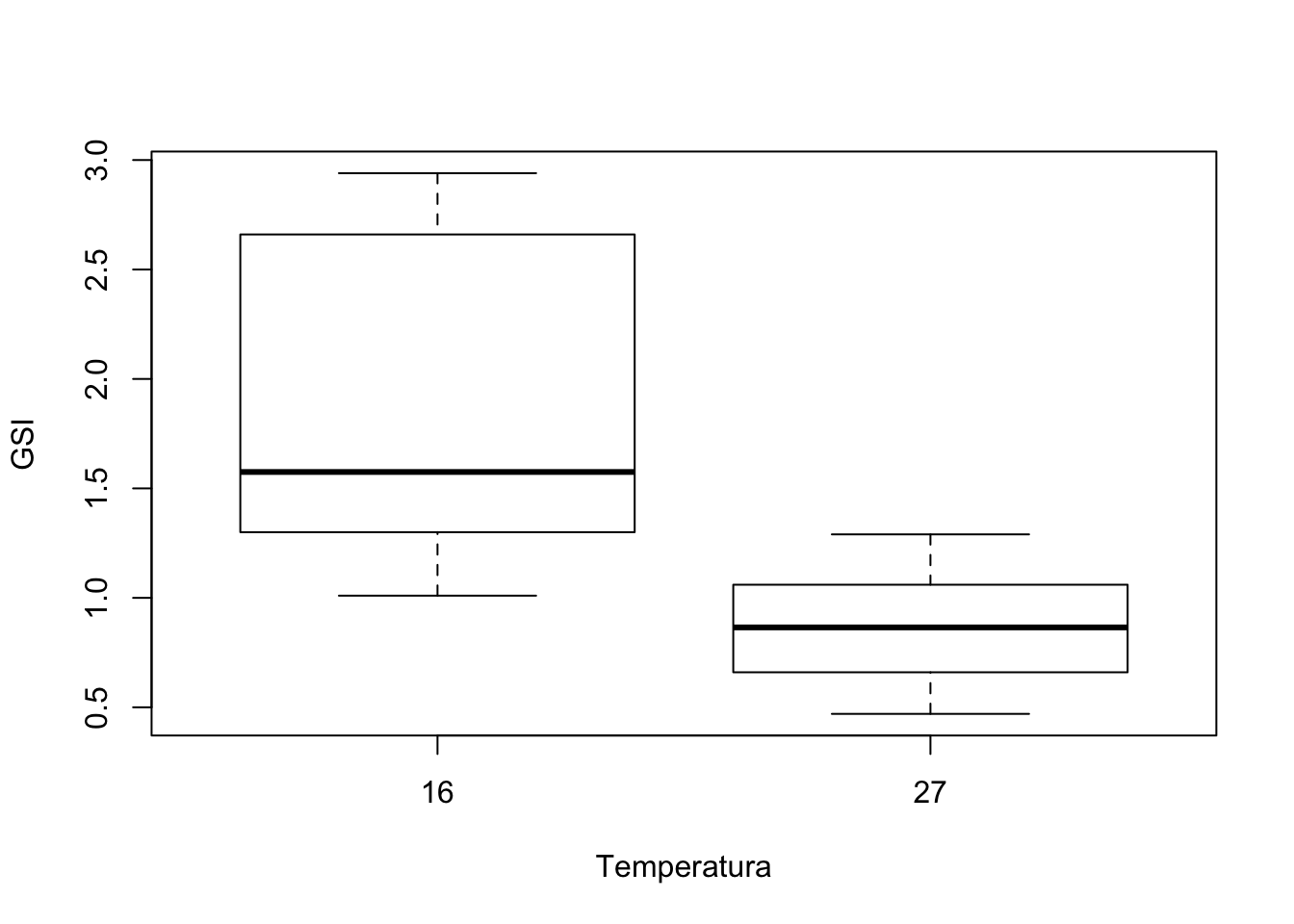
Si hacemos el boxplot de la variable GSI según la combinación de los dos factores, fotoperíodo y temperatura, obtenemos:
boxplot(GSI ~ fotoperiodos+temperatura, data = tabla.datos.GSI,
xlab="Combinación Fotoperíodo.Temperatura",ylab="GSI")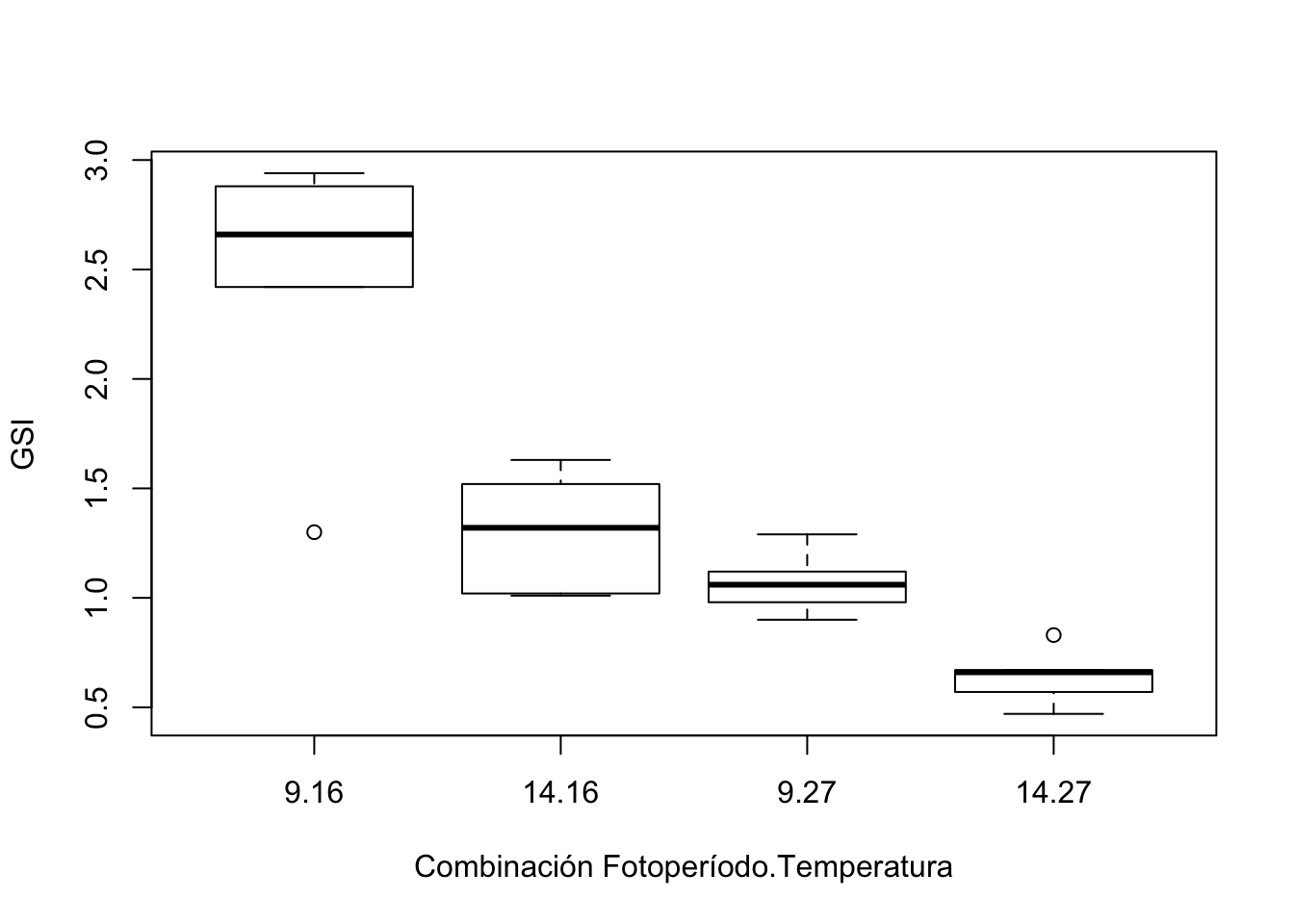
Parece que sí hay diferencias en la variable GSI según el fotoperíodo, según la temperatura y según la combinación fotoperíodo/temperatura.
El contraste ANOVA de dos vías para los datos de nuestro ejemplo se realiza de la forma siguiente en R:
## Df Sum Sq Mean Sq F value Pr(>F)
## fotoperiodos 1 3.08 3.08 21.70 0.00026 ***
## temperatura 1 5.15 5.15 36.29 0.000018 ***
## fotoperiodos:temperatura 1 0.63 0.63 4.44 0.05127 .
## Residuals 16 2.27 0.14
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Obtenemos los mismos resultados que hemos obtenido anteriormente.
Observamos que falta la fila de los tratamientos.
Para realizar el contraste de los tratamientos en R, hacemos lo siguiente:
## Df Sum Sq Mean Sq F value Pr(>F)
## fotoperiodos:temperatura 3 8.86 2.954 20.8 0.0000091 ***
## Residuals 16 2.27 0.142
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 17.6.12 Gráficos de interacción
El gráfico de interacción entre los dos factores consiste en unir mediante segmentos los valores medios que toma la variable que comparamos X para cada factor en los que hemos segregado dicha variable.
Si no hay ninguna interacción entre los factores, los segmentos anteriores son paralelos. Cuanto más alejados del paralelismo estén dichos segmentos, más evidencia de interacción existe entre estos dos factores.
Para realizar un gráfico de interacción en R se usa la función interaction.plot:
donde F1 es el factor que dibujamos en el eje de abscisas o eje X y F2 es el otro factor usado para dibujar los segmentos.
Para dibujar el gráfico de interacción entre factores, vamos a crear primero las tres variables siguientes:
GSI=tabla.datos.GSI$GSI
fotoperiodos=tabla.datos.GSI$fotoperiodos
temperatura=tabla.datos.GSI$temperaturaPara dibujar el gráfico de interacción del fotoperíodo según la temperatura, hacemos lo siguiente:

Para dibujar el gráfico de interacción de la temperatura según el fotoperíodo, hacemos lo siguiente:
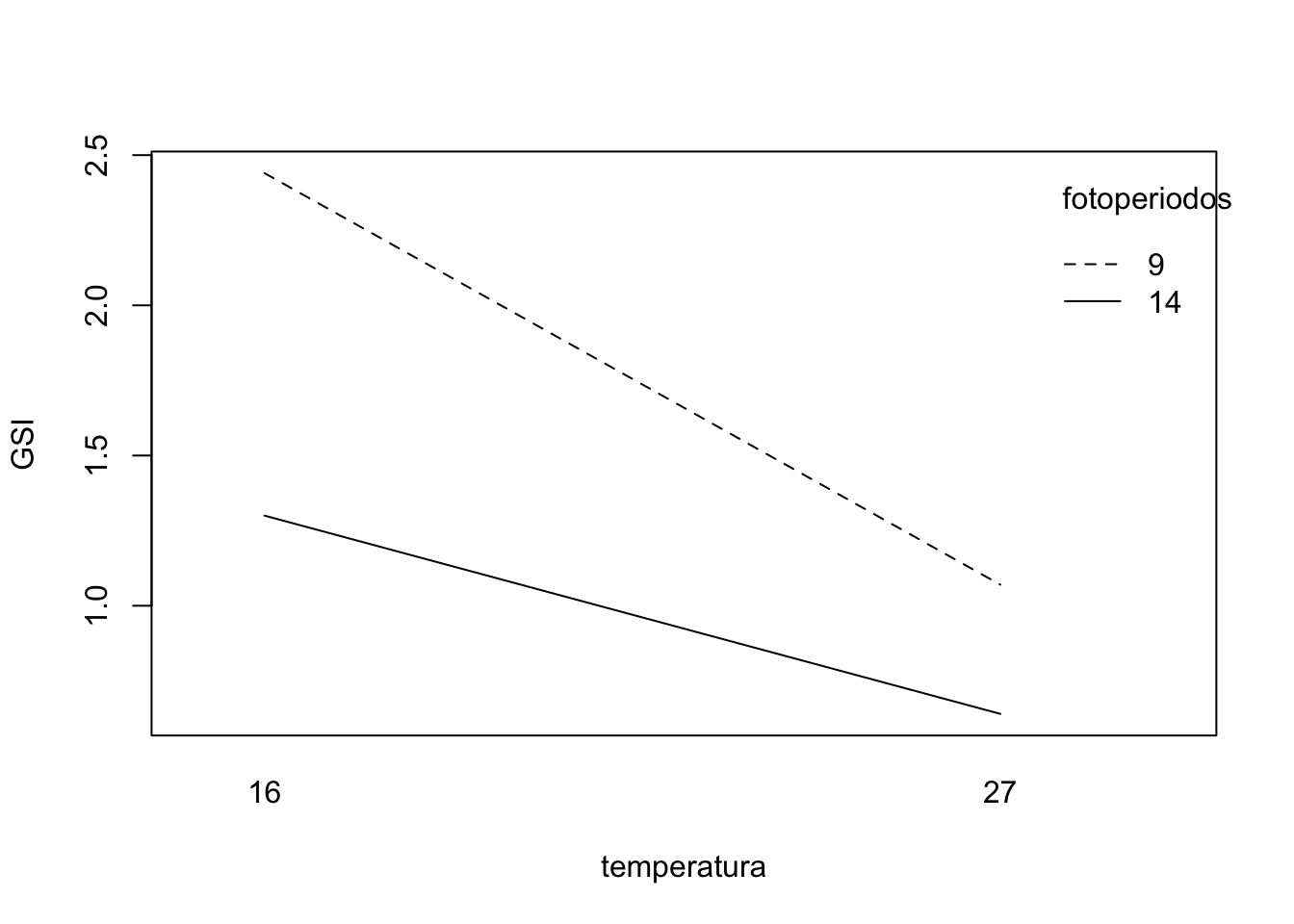
Vemos que las rectas anteriores no son paralelas pero tampoco “parecen” estar demasiado lejos del paralelismo.
De ahí el p-valor obtenido en la zona de penumbra.
7.7 Guía rápida
summary(aov(X ~ F))nos la tabla ANOVA de un factor cuando contrastamos las medias de las subpoblaciones de la variableXsegregada según los niveles de la variable factorF.pairwise.t.test(X,F,p.adjust.method = ...)nos realiza la comparación por parejas de niveles del factorFentre las medias de las subpoblaciones de la variableX. El parámetrop.adjust.methodpuede ser:none: no hace ajuste alguno,bonferroni: realiza el ajuste del método de Bonferroni: multiplicar el p-valor del contraste por el número de comparaciones llevadas a cabo, \(\binom{k}{2}\).holm: realiza el ajuste del método de Holm.
duncan.test(aov,"factor",group=...)$sufijodel paqueteagricolae. Realiza el test de Duncan de comparación por parejas de niveles del factorFentre las medias de las poblaciones de la variableX, donde:aoves el resultado del ANOVA de partida,- el
factores el factor del ANOVA, grouppuede serTRUEoFALSEdependiendo de cómo queremos ver el resultado,- el
sufixesgroupsigroup=TRUEycomparisonsigroup=FALSE.
TukeyHSD(aov(X~F)): realiza el test de Tukey de comparación por parejas de niveles del factorFentre las medias de las subpoblaciones de la variableX.bartlett.test(X~F): realiza el test de Bartlett para ver si las varianzas de las subpoblaciones de la variableXsegregada según los niveles de la variable factorFson iguales o no.summary(aov(X ~ Tr+Bl)): nos la tabla ANOVA de bloques completos aleatorios cuando contrastamos las medias de las subpoblaciones de la variableXsegregada según los niveles de la variable factorTr(tratamientos) donde los valores de cada vector de tratamientos está emparejada según la variable factorBl(bloques).summary(aov(X ~ A*B)): nos da la tabla ANOVA de dos factores cuando cuando contrastamos las medias de las subpoblaciones de la variableXsegregada según los niveles de las variables factorAyB.interaction.plot(F1,F2,X): nos da el gráfico de interacción de los niveles del factorF1según los niveles de factorF2yXes la variable cuyas medias contrastamos en el ANOVA.